ГОСТ Р ИСО/МЭК 19794-14-2017
Группа П85
НАЦИОНАЛЬНЫЙ СТАНДАРТ РОССИЙСКОЙ ФЕДЕРАЦИИ
Информационные технологии
БИОМЕТРИЯ
Форматы обмена биометрическими данными
Часть 14
Данные ДНК
Information technology. Biometrics. Biometric data interchange formats. Part 14. DNA data
ОКС 35.040
Дата введения 2018-12-01
Предисловие
1 ПОДГОТОВЛЕН Научно-исследовательским и испытательным центром биометрической техники Московского государственного технического университета имени Н.Э.Баумана (НИИЦ БТ МГТУ им.Н.Э.Баумана) и федеральным государственным унитарным предприятием "Всероссийский научно-исследовательский институт стандартизации и сертификации в машиностроении" (ВНИИНМАШ) на основе собственного перевода на русский язык англоязычной версии стандарта, указанного в пункте 4
2 ВНЕСЕН Техническим комитетом по стандартизации ТК 098 "Биометрия и биомониторинг"
3 УТВЕРЖДЕН И ВВЕДЕН В ДЕЙСТВИЕ Приказом Федерального агентства по техническому регулированию и метрологии от 9 июня 2017 г. N 528-ст
4 Настоящий стандарт идентичен международному стандарту ИСО/МЭК 19794-14:2013* "Информационные технологии. Форматы обмена биометрическими данными. Часть 14. Данные ДНК" (ISO/IEC 19794-14:2013 "Information Technology - Biometric data interchange Formats - Part 14: DNA data", IDT).
________________
* Доступ к международным и зарубежным документам, упомянутым в тексте, можно получить, обратившись в Службу поддержки пользователей. - .
Международный стандарт разработан подкомитетом ИСО/МЭК СТК 1/ПК 37 "Биометрия" совместного технического комитета по стандартизации ИСО/МЭК СТК 1 "Информационные технологии" Международной организации по стандартизации (ИСО) и Международной электротехнической комиссии (МЭК).
Наименование настоящего стандарта изменено относительно наименования указанного международного стандарта для приведения в соответствие с ГОСТ Р 1.5-2012 (пункт 3.5).
При применении настоящего стандарта рекомендуется использовать вместо ссылочных международных стандартов соответствующие им национальные стандарты, сведения о которых приведены в дополнительном приложении ДА
5 ВВЕДЕН ВПЕРВЫЕ
6 ПЕРЕИЗДАНИЕ. Январь 2019 г.
7 Некоторые элементы настоящего стандарта могут быть объектами патентных прав. ИСО и МЭК не несут ответственности за установление подлинности каких-либо или всех таких патентных прав
Правила применения настоящего стандарта установлены в статье 26 Федерального закона от 29 июня 2015 г. N 162-ФЗ "О стандартизации в Российской Федерации". Информация об изменениях к настоящему стандарту публикуется в ежегодном (по состоянию на 1 января текущего года) информационном указателе "Национальные стандарты", а официальный текст изменений и поправок - в ежемесячном информационном указателе "Национальные стандарты". В случае пересмотра (замены) или отмены настоящего стандарта соответствующее уведомление будет опубликовано в ближайшем выпуске ежемесячного информационного указателя "Национальные стандарты". Соответствующая информация, уведомление и тексты размещаются также в информационной системе общего пользования - на официальном сайте Федерального агентства по техническому регулированию и метрологии в сети Интернет (www.gost.ru)
Введение
За последние 20 лет судебная молекулярная генетика из быстро развивающейся области знаний с меняющимися технологиями развилась в общепризнанную судебную науку.
Судебная генетика, использующая анализ дезоксирибонуклеиновой кислоты (ДНК), включает ряд важных областей применения. Примером является исследование биологических следов для получения доказательства присутствия предполагаемого преступника на месте совершения преступления путем сравнения генетических профилей образцов человеческого происхождения с места преступления с генетическими профилями баз данных ДНК правоохранительных органов. Областями применения также являются идентификация трупов неизвестных лиц, в т.ч. жертв массовых бедствий, иммиграция, установление отцовства.
Целью настоящего стандарта является определение формата для обмена идентификационными данными ДНК человека. Настоящий стандарт определяет атрибуты ДНК и формат записи для обмена данными ДНК, а также включает в себя пример записи данных ДНК.
Настоящий стандарт основан на данных ДНК, получаемых стандартизованными и наиболее часто используемыми методами судебно-медицинского ДНК-типирования, например КТП-анализ.
_______________
КТП - короткий тандемный повтор (Short tandem repeat (STR)).
Настоящий формат обмена данными ДНК предназначен для обмена данными ДНК в различных системах и не накладывает каких-либо ограничений на использование конкретных систем и методов ДНК-типирования.
Существующие форматы обмена данными ДНК, использованные при подготовке настоящего стандарта, перечислены в качестве ссылок.
Типовые системы профилирования используют некодируемые участки ДНК ("мусорная ДНК"). Кодируемые участки ДНК были целенаправленно исключены для обеспечения конфиденциальности и соблюдения гражданских прав донора. Тем не менее, национальное законодательство о защите персональных данных может определять особые гарантии безопасности, такие как шифрование передаваемых данных и/или хранимых данных и другие методы.
1 Область применения
Настоящий стандарт устанавливает формат обмена данными ДНК для технологий идентификации или верификации личности.
Настоящий стандарт обеспечивает возможность обмена и использования данных ДНК-профиля для сравнения (с учетом правил конфиденциальности) с данными ДНК-профиля, полученными любой другой системой с совместимыми методами ДНК-профилирования и форматом обмена биометрическими данными, соответствующему настоящему стандарту.
Настоящий стандарт распространяется на действующие в судебной медицине методы ДНК- профилирования или ДНК-типирования, основанные на коротких тандемных повторах (КТП), включая КТП на Y-хромосоме (Y-КТП) и на митохондриальной ДНК.
Настоящий формат данных подготовлен с учетом тенденции по сокращению участия человека в анализе (регистрации и сравнении) ДНК. С учетом требований к формату данных для автоматизированных методов анализа ДНК, настоящий стандарт определяет формат как для обработанных, так и необработанных (электрофоретических) данных ДНК. Для определения обмена данными ДНК используется кодировка расширяемого языка разметки (Extensible Mark-up Language, XML). Пример XML-схемы данных ДНК представлен в приложении В.
Настоящий стандарт не предназначен для любых других целей, кроме как обмен данными ДНК для биометрической идентификации или верификации личности, в частности, не предназначен для обмена медицинской или другой информацией о здоровье.
2 Соответствие
Приложения, заявленные как соответствующие настоящему стандарту, должны демонстрировать представление биометрических данных ДНК в соответствии с настоящим стандартом. Минимальные требования к соответствию стандарту включают способность передачи (обмена) и извлечения совместимой биометрической информации ДНК.
3 Нормативные ссылки
В настоящем стандарте использованы нормативные ссылки на следующие международные стандарты. Для датированных ссылок применяют только указанное издание ссылочного стандарта.
ISO/IEC 19794-1:2011 Information technology - Biometric data interchange formats - Part 1: Framework (Информационные технологии. Форматы обмена биометрическими данными. Часть 1. Структура)
ISO/IEC 19794-1:2011/Amd.2 Information technology - Biometric data interchange formats - Part 1: Framework - Amendment 2: Framework for XML encoding (Информационные технологии. Форматы обмена биометрическими данными. Часть 1. Структура. Изменение 2. Структура XML кодирования).
4 Термины и определения
В настоящем стандарте применены термины по ИСО/МЭК 19794-1:2011, а также следующие термины с соответствующими определениями:
4.1 аллель (allele): Одна из возможных альтернативных форм последовательности ДНК, находящейся на местоположении определенного гена.
4.2 хромосома (chromosome): Структура клетки, которая переносит генетический материал в виде линейной цепи ДНК.
Примечание - В человеческой клетке содержится 23 пары хромосом.
4.3 дезоксирибонуклеиновая кислота; ДНК (deoxyribo nucleic acid, DNA): Сложная молекула, находящаяся практически в каждой клетке тела и несущая генетическую информацию от одного поколения к другому.
4.4 ДНК-профилирование или ДНК-типирование (DNA profiling or typing): Метод, используемый учеными для различения индивидов путем изучения отличий их ДНК.
________________
В биометрии термин "индивид" относится только к человеку.
4.5 локус (locus): Уникальное физическое расположение на молекуле ДНК (во множественном числе - локусы).
4.6 митохондриальная ДНК; мтДНК (mitochondrial DNA, mtDNA): Небольшие кольцевые молекулы ДНК, расположенные в структурах, используемых для получения энергии в клетке (митохондриях).
Примечание - Небольшой размер и большое количество делают их особенно эффективными при исследовании малого количества биологического материала или в случае его сильного повреждения. Митохондриальная ДНК может быть использована для отслеживания передачи материнской линии, поскольку наследование мтДНК происходит только по материнской линии.
4.7 вероятность дискриминации неродственных индивидов (power of discrimination): Эффективность использования генетического маркера или набора маркеров для различения любых двух случайно выбранных людей.
4.8 короткий тандемный повтор; КТП (short tandem repeat, STR): Короткие последовательности ДНК, которые многократно повторяются при прямом наследовании.
Примечание - Число коротких повторов может широко варьироваться у разных индивидов, высокий уровень изменчивости делает КТП особенно эффективным для различения индивидов.
4.9 Y-хромосома (Y chromosome): Организованная структура молекулы ДНК, содержащая специфичную только для мужчин ДНК.
4.10 Y-КТП (Y-STR): Участки КТП, находящиеся в специфичной только для мужчин ДНК хромосом Y.
Примечание - Y-КТП могут быть использованы для отслеживания передачи отцовской линии, поскольку они встречаются только у мужчин, и наследование происходит только по отцовской линии.
4.11 электрофоретические данные (electrophoretic data): Необработанные данные на выходе системы профилирования, которые используются для измерения числа повторов аллелей в определенных локусах.
4.12 мобильный модуль обработки (mobile processing unit): Мобильная, полностью функциональная лаборатория ДНК.
4.13 быстрый модуль ДНК (rapid DNA unit): Автономное устройство, которое автоматизирует все процессы анализа ДНК и быстро производит ДНК-профиль (например, 1 ч).
5 Сокращения
В настоящем стандарте применены следующие сокращения:
ДНК - дезоксирибонуклеиновая кислота;
мтДНК - митохондриальная ДНК;
КТП - короткий тандемный повтор;
Y-КТП - КТП Y-хромосомы.
6 Структура формата записи данных ДНК
6.1 Общие положения
Настоящий стандарт определяет структуру записи данных ДНК. Формат данных должен содержать идентификационные данные ДНК. В соответствии с требованиями ИСО/МЭК 19794-1, запись данных ДНК должна быть определена как ЗОБД или включена в ББД
структуры, совместимой с ЕСФОБД (ЗБИ
).
________________
ЗОБД - Запись для обмена биометрическими данными.
ББД - Блок биометрических данных.
ЗБИ - Запись биометрической информации.
Элементы записи сгруппированы по трем структурам данных (поля, блоки и запись). Элемент "поле" обозначает простую структуру для хранения данных. Поля могут быть двух типов: простое и составное. Простое поле содержит только один объект данных, составное поле содержит одно или более полей, которые, в свою очередь, могут быть простыми или составными. Одно или более полей могут быть сгруппированы в блок данных. Сегмент, состоящий из нескольких элементов с уникальными названиями (полей или блоков данных) формируют запись данных.
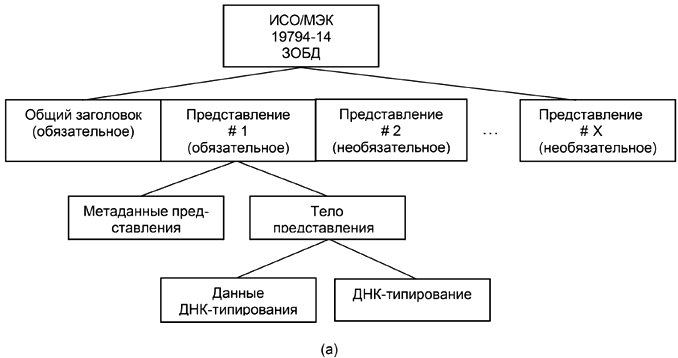
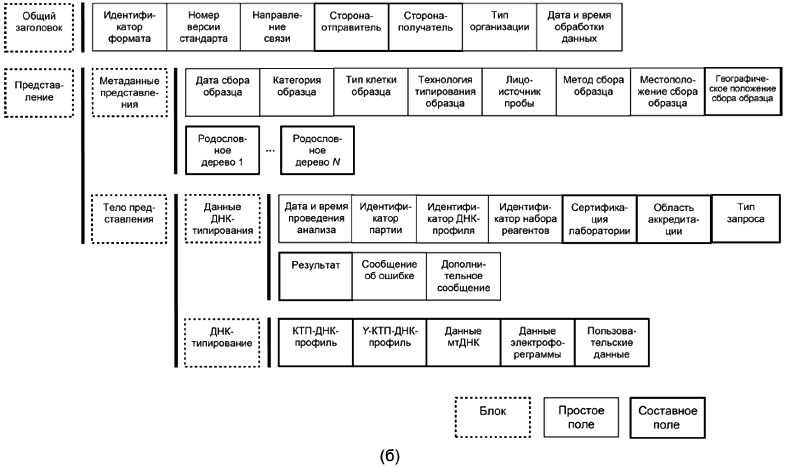
Структура формата записи данных ДНК и порядок расположения блоков и полей записи данных ДНК представлен на рисунке 1.
|
Рисунок 1 - Структура формата записи данных ДНК (а) и порядок расположения блоков и полей записи данных ДНК (б)
|
Рисунок 1, лист 2
6.2 Соглашения в отношении данных
6.2.1 Значение поля "Неизвестно"
Значение поля с идентификатором "Неизвестно" должно быть использовано для обозначения того, что информация для кодирования в данном поле еще не определена.
6.2.2 XML-кодирование
Структура XML-кодирования рассмотрена в ИСО/МЭК 19794-1:2011 (Изм.2). Схема XML- кодирования данных ДНК, представленная в приложении В настоящего стандарта, соответствует совместно применяемой спецификации для структуры XML (ИСО/МЭК 19794-1:2011 (Изм.2)).
6.3 Заголовок ЕСФОБД (CBEFF header)
Для записи данных ДНК должна быть использована структура ЗОБД из одного обязательного блока "Общий заголовок" (General header) и одного или более блоков "Представление" (Representation).
ЗОБД, соответствующая формату записи данных ДНК, может быть включена в ББД (BDB) формата ведущей организации ЕСФОБД (CBEFF patron format) в соответствии с ИСО/МЭК 19785-1:2004. При использовании заголовка ЕСФОБД должны быть удовлетворены следующие требования:
- в формате ведущей организации ЕСФОБД должны быть определены элементы "CBEFF_BDB_ format_owner" (владелец формата ББД ЕСФОБД) и "CBEFF_BDB_format_type" (тип формата ББД ЕСФОБД) как обязательные элементы заголовка ЕСФОБД;
- значение элемента "CBEFF_BDB_format_owner" (владелец формата ББД ЕСФОБД) должно определять идентификатор организации-участника ЕСФОБД (CBEFF biometric organization identifier), присвоенный регистрационным органом ЕСФОБД для ИСО/МЭК СТК 1/ПК 37. Значение элемента должно быть 0x0101 (16 бит);
- значение элемента "CBEFF_BDB_format_type" (тип формата ББД ЕСФОБД) должно определять идентификатор типа формата ББД ЕСФОБД, присвоенный ИСО/МЭК СТК 1/ПК 37 для формата записи данных ДНК. Значение элемента должно быть 0x0008 (16 бит);
- полная информация, необходимая для кодирования заголовка ЕСФОБД (CBEFF header), приведена в ИСО МЭК 19794-1.
6.4 Структура записи данных ДНК
6.4.1 Структура блока "Общий заголовок"
Блок "Общий заголовок" записи данных ДНК состоит из 7 полей (таблица 1). Структура блока "Общий заголовок представлена в таблице 1. Поля, перечисленные в первом столбце таблицы 1, рассмотрены более подробно в 6.4.1.1-6.4.1.7.
Таблица 1 - Структура блока "Общий заголовок"
Поле | Тип | Допустимое | Обязательное/ |
Идентификатор формата (Format identifier) | string | "DNA" | Обязательное |
Номер версии стандарта (Version number) | VersionType | Номер версии = 3, номер поправки = 0 | Обязательное |
Направление связи (Communication direction) | string | "Request", "Answer" | Обязательное |
Сторона-отправитель (Sending party) | PartyType | - | Обязательное |
Сторона-получатель (Receiving party) | PartyType | - | Обязательное |
Тип организации (Entity type) | string | "G", "GM", "GR", "I", "IМ", "IR", "О", "ОМ", "OR", "U", "UM", "UR" | Обязательное |
Дата и время обработки данных (Date and time of data processing) | dateTime | - | Обязательное |
| |||
6.4.1.1 Поле "Идентификатор формата"
Поле "Идентификатор формата" для данных ДНК должно иметь значение "DNA" с типом данных string.
6.4.1.2 Поле "Номер версии стандарта"
Поле "Номер версии стандарта" должно содержать номер версии стандарта и номер поправки или изменения редакции. Формат значения поля должен соответствовать ИСО/МЭК 19794-1:2011 (Изм.2). Номер версии настоящего стандарта должен иметь значение 3 для номера версии стандарта и значение 0 для номера поправки или изменения редакции.
6.4.1.3 Поле "Направление связи"
Поле "Направление связи" должно определять, является сообщение запросом или ответом на запрос (таблица 2). Тип данных поля должен быть string.
Таблица 2 - Поле "Направление связи"
Описание | Значение |
Запрос | "Request" |
Ответ | "Answer" |
6.4.1.4 Поле "Сторона-отправитель"
Поле "Сторона-отправитель" должно иметь тип данных PartyType и должно состоять из полей "Код государства" (Nationality code), "Наименование организации" (Name of the entity) и "ФИО должностного лица" (Name of the person) (в качестве должностного лица указывается отправитель) (таблица 3). Поле "Сторона-отправитель" является составным.
Таблица 3 - Тип данных PartyType
Поле | Тип данных | Описание |
Код государства | string | Код в соответствии с ИСО/МЭК 3166-2 |
Наименование организации | string | Наименование организации |
ФИО должностного лица | string | Ф.И.О. отправителя/получателя |
6.4.1.5 Поле "Сторона-получатель"
Поле "Сторона-получатель" должно иметь тип данных PartyType и должно состоять из полей "Код государства", "Наименование организации" и "ФИО должностного лица" (в качестве должностного лица указывается получатель) (таблица 3). Поле "Сторона-получатель" является составным.
6.4.1.6 Поле "Тип организации"
В качестве типа организации могут указываться "Государственная лаборатория" (G) (Government lab), "Коммерческая лаборатория" (I) (Industry lab), "Другая лаборатория" (О) (Other lab) или "Неизвестная лаборатория" (U) (Unknown lab). К типу организации может добавляться описание "Быстрый модуль ДНК" (R) (Rapid DNA unit) или "Мобильный модуль обработки" (М) (Mobile processing unit). Таким образом, поле "Тип организации" должно иметь тип данных string и значение из допустимых: "G", "GM", "GR", "I", "IМ", "IR", "О", "ОМ", "OR", "U", "UM", "UR".
6.4.1.7 Поле "Дата и время обработки данных"
В данном поле должны быть представлены дата и время обмена биометрическими данными по Гринвичу (универсальное глобальное время). Для любых абсолютных значений времени должен быть использован формат в соответствии с ИСО/МЭК 19794-1:2011 (Изм.2). Данное поле определяет дату и время обработки биометрических данных. Поле должно иметь встроенный в ХМL тип данных dateTime.
6.4.2 Блок "Метаданные представления" (Representation metadata)
Для каждого профиля должно* быть блок "Представление" (Representation), который состоит из блока "Метаданные представления" и блока "Тело представления" (Representation body). В блоке "Метаданные представления" представлены метаданные о данных обмена. Структура блока "Метаданные представления" представлена в таблице 4.
________________
* Текст документа соответствует оригиналу. - .
Таблица 4 - Структура блока "Метаданные представления"
Поле | Тип | Допустимое | Обязательное/ |
Дата сбора образца (Sample collection date) | dateTime | - | Необязательное |
Категория образца (Sample category) | string | См. таблицу 5 | Обязательное |
Тип клетки образца (Sample cellular type) | string | См. таблицу 6 | Обязательное |
Технология типирования образца (Sample typing technology) | string | См. таблицу 7 | Обязательное |
Лицо-источник пробы (Specimen contributor) | string | "Known" или "Unknown" | Обязательное |
Метод сбора образца (Sample collection method) | string | - | Необязательное |
Местоположение сбора образца (Sample collection location) | string | - | Необязательное |
Географическое положение сбора образца (Sample collection geo-location) | GeoLocationType | - | Необязательное |
Родословное дерево (Pedigree tree) | PedigreeTreeType | - | Необязательное |
6.4.2.1 Поле "Дата сбора образца"
В поле "Дата сбора образца" должна быть указана дата, когда был собран образец, по Гринвичу (универсальное глобальное время). Для любых абсолютных значений времени должен быть использован формат в соответствии с ИСО/МЭК 19794-1:2011 (Изм.2). Поле должно иметь встроенный в XML тип данных "dateTime".
6.4.2.2 Поле "Категория образца"
Поле "Категория образца" должно определять категорию, к которой принадлежит образец ДНК (таблица 5). Тип данных поля должен быть string.
Таблица 5 - Поле "Категория образца"
Значение |
"Arrestee" (Арестованное лицо) |
"Claimed Biological Child" (Заявленный биологический ребенок) |
"Claimed Biological Father" (Заявленный биологический отец) |
"Claimed Biological Mother" (Заявленная биологическая мать) |
"Claimed Biological Sibling" (Заявленный/ая биологический/ая родной/ая брат/сестра) |
"Claimed Biological Spouse" (Заявленный биологический супруг) |
"Actual Biological Child" (Действительный биологический ребенок) |
"Actual Biological Father" (Действительный биологический отец) |
"Actual Biological Mother" (Действительная биологическая мать) |
"Actual Biological Sibling" (Действительный/ая биологический/ая родной/ая брат/сестра |
"Actual Biological Spouse" (Действительный биологический супруг) |
"Adoptive Biological Child" (Приемный биологический ребенок) |
"Adoptive Biological Father" (Приемный биологический отец) |
"Adoptive Biological Mother" (Приемная биологическая мать) |
"Adoptive Biological Sibling" (Приемная биологический/ая родной/ая брат/сестра) |
"Adoptive Biological Spouse" (Приемный биологический супруг) |
"Convicted Offender" (Осужденное лицо) |
"Forensic, Unknown" (Криминалистика, неизвестное лицо) |
"Insurgent" (Боевик) |
"Known Suspected Terrorist" (Известное лицо, подозреваемое в терроризме) |
"Maternal relative" (Родственник по материнской линии) |
"Missing person" (Лицо, пропавшее без вести) |
"Paternal Relative" (Родственник по отцовской линии) |
"Suspect, Known" (Известное подозреваемое лицо) |
"Unidentified Living" (Неопознанный живой человек) |
"Unidentified Dead" (Труп неопознанного человека) |
"Victim, Known" (Жертва, известное лицо) |
"Detainee" (Задержанное лицо) |
"Other" (Другое) |
"Unspecified" (Не определено) |
Примечание - Некоторые термины, перечисленные в данной таблице, могут иметь разный смысл в различных правовых системах (например, detainee (задержанное лицо), arrestee (арестованное лицо), convicted offender (осужденное лицо)).
6.4.2.3 Поле "Тип клетки образца"
Поле "Тип клетки образца" должно определять источник типа клетки, из которого получен образец (таблица 6). Тип данных поля должен быть string.
Таблица 6 - Поле "Тип клетки образца"
Значение |
"Blood" (Кровь) |
"Воnе" (Кость) |
"Buccal Cell" (Клетка буквального эпителия) |
"Commingled Biological Material" (Смешанный биологический материал) |
"Hair" (Волосы) |
"Saliva" (Слюна) |
"Semen" (Сперма) |
"Skin" (Кожа) |
"Sweat/Fingerprint" (Пот/отпечаток пальца) |
"Tissue" (Носовой платок или салфетка) |
"Tooth (including Pulp)" (Зуб (включая пульпу)) |
"Other" (Другое) |
"Unknown" (Неизвестно) |
"Unspecified" (Не определено) |
6.4.2.4 Поле "Технология типирования образца"
Поле "Технология типирования образца" должно определять технологию, используемую для ДНК-типирования (таблица 7). Тип данных поля должен быть string.
Таблица 7 - Поле "Технология типирования образца"
Значение |
"STR"(КТП) |
"Y-STR" (Y-КТП) |
"mtDNA" (мтДНК) |
"Electropherogram" (Электрофореграмма) |
"User Defined Typing" (Типирование, определенное пользователем) |
6.4.2.5 Поле "Лицо-источник пробы"
Значение поля "Лицо-источник пробы" определяет, известна ли личность источника пробы или нет (таблица 8). Значение поля должно иметь тип string.
Таблица 8 - Поле "Лицо-источник пробы"
________________
В ИСО/МЭК 19794-14:2013 допущена опечатка - указано название таблицы 8 "Sample source indicator" вместо "Specimen conductor".
Описание | Значение |
Лицо-источник пробы известно | "Known" (Известно) |
Лицо-источник пробы неизвестно | "Unknown" (Неизвестно) |
Примечание - Данное поле целесообразно использовать в случае неопознанного живого человека, когда сбор образца проводится непосредственно с человека. В случае лица, пропавшего без вести, образец может не принадлежать человеку, объявленному пропавшим.
6.4.2.6 Поле "Метод сбора образца"
Поле "Метод сбора образца" должно определять описание метода сбора образца. Значение поля должно иметь тип string.
Примечание - Например, образец "волосы" может быть получен непосредственно с человека или обнаружен на месте преступления.
6.4.2.7 Поле "Местоположение сбора образца"
Поля "Местоположение сбора образца" должно определять местоположение, где был собран образец. Это строковое описание, например, дома, здания и/или почтовые адреса и т.д.
6.4.2.8 Поле "Географическое положение сбора образца"
Поле "Географическое положение сбора образца" должно содержать значения GPS-координат места сбора образца (таблица 9). Такие данные позволяют обрабатывать данные массовых бедствий. Значение поля должно состоять из двух значений типа float. Первое значение - географическая широта, второе значение - географическая долгота в WGS-84 (Мировая геодезическая система, World Geodetic System). Поле "Географическое положение сбора образца" является составным, структура данного поля представлена в таблице 9.
Таблица 9 - Структура поля "Географическое положение сбора образца" (тип данных - GeoLocationType)
Поле | Тип данных | Примечание |
Географическая широта (Lattitude) | float | + для северного полушария, |
Географическая долгота (Longitude) | float | + для восточного полушария, |
6.4.2.9 Поле "Родословное дерево"
Поле "Родословное дерево" должно повторяться нужное число раз для построения полного дерева (таблицы 10-12). В родословном дереве должен быть указан минимум один член. Генетические данные должны соответствовать представленной информации. Родословные деревья в значительной степени способствуют идентификации лиц, пропавших без вести, или идентификации жертв массовых бедствий. Каждое родословное дерево может иметь одно или более неустановленных узлов, отображающих неизвестное лицо. Поле "Родословное дерево" является составным.
Таблица 10 - Поле "Родословное дерево"
Поле | Тип данных | Допустимое | Примечание |
Родословное дерево 1 (Pedigree Tree 1) | PdgrTreeType | - | Повторяется число раз, равное числу родословных деревьев |
Таблица 11 - Поле "Родословное дерево" (тип данных - PdgrTreeType)
Поле | Тип данных | Допустимое | Примечание |
Идентификатор родословного дерева (Pedigree ID) | string | - | Уникальный идентификатор родословного дерева |
Член родословного дерева 1 (Pedigree Member 1) | PdgrMbrType | - | Повторяется число раз, равное числу членов родословного дерева |
Таблица 12 - Поле "Член родословного дерева" (тип данных - PdgrMbrType)
Поле | Тип | Допустимое | Примечание |
Идентификатор члена родословного дерева (Pedigree member ID) | integer | - | Уникальный идентификатор родословного дерева в формате целого числа |
Идентификатор образца (Specimen ID) | string | Длина | Если образец связан с родословной узла, то идентификатор образца должен быть определен и включен в раздел образца файла импорта родословного дерева. Узлы родословного дерева, в которых имеются образцы, считаются "заполненными". Если для узла родословного дерева не указан ни один образец, узел считается "незаполненным" |
Идентификатор матери (Mother ID) | integer | - | Если указан идентификатор матери, то должен быть указан идентификатор отца |
Идентификатор отца (Father ID) | integer | - | Если указан идентификатор отца, то должен быть указан идентификатор матери |
Статус члена родословного дерева (Pedigree member status) | string | "Known" (Известен) или "Unknown" (Неизвестен) | Известен или неизвестен |
Пол (Gender) | string | "Male" (Мужской) или "Female" (Женский) | Мужской или женский |
6.4.3 Блок "Тело представления"
6.4.3.1 Блок "Данные ДНК-типирования"
Структура блока "Данные ДНК-типирования" представлена в таблице 13.
Таблица 13 - Структура блока "Данные ДНК-типирования"
Поле | Тип | Допустимое | Обязательное/ |
Дата и время проведения анализа (Date and time of analysis) | dateTime | - | Необязательное |
Идентификатор партии (Batch ID) | string | - | Обязательное |
Идентификатор ДНК-профиля (DNA profile ID) | string | - | Обязательное |
Идентификатор набора реагентов (Kit ID) | string | - | Обязательное |
Сертификация лаборатории (Lab certification) | LabCertType | - | Обязательное |
Область аккредитации (Scope of accreditation) | SOAType | - | Обязательное |
Тип запроса (Request type) | string | - | Обязательное, если значение в поле "Направление связи" равно "R" (Request, запрос). Иначе - необязательное |
Результат (Result) | string | - | Обязательное, если значение в поле "Направление связи" равно "R" (Request, запрос). Иначе - необязательное |
Сообщение об ошибке (Error message) | string | - | Необязательное |
Дополнительное сообщение (Supplementary message) | string | - | Необязательное |
6.4.3.1.1 Поле "Дата и время проведения анализа"
В поле "Дата и время проведения анализа" должны быть представлены дата и время проведения анализа по Гринвичу (универсальное глобальное время). Для любых абсолютных значений времени должен быть использован формат в соответствии с ИСО/МЭК 19794-1:2011 (Изм.2). Поле должно иметь встроенный в ХМL тип данных dateTime (xs:dateTime).
6.4.3.1.2 Поле "Идентификатор партии"
В поле "Идентификатор партии" должен быть указан идентификатор партии, в которой проводится ДНК-типирование. Поле должно иметь тип данных string.
Примечание - Если идентификатор партии неизвестен, то значение поля должно быть "Unknown" (Неизвестно).
6.4.3.1.3 Поле "Идентификатор ДНК-профиля"
В поле "Идентификатор ДНК-профиля" должен быть указан уникальный идентификатор ДНК-профиля, назначенный стороной-отправителем. Поле должно иметь тип данных string.
6.4.3.1.4 Поле "Идентификатор набора реагентов"
В поле "Идентификатор набора реагентов" должен быть указан идентификатор используемого набора реагентов. Поле должно иметь тип данных string. Примеры наборов реагентов для ДНК-анализа приведены в приложении С.
Примечание - Если идентификатор набора реагентов неизвестен, значение поля должно быть равно "Unknown" (Неизвестно).
6.4.3.1.5 Поле "Сертификация лаборатории"
Поле "Сертификация лаборатории" должно определять статус качества лаборатории. Поле "Сертификация лаборатории" является составным. Структура поля "Сертификация лаборатории" представлена в таблице 14. Допустимые значения поля "Сертификация лаборатории" представлены в таблице 15. Лаборатория может иметь несколько валидаций (сертификаций).
________________
В ИСО/МЭК 19794-14:2013 допущена опечатка - предложение "Лаборатория может иметь несколько валидаций (сертификаций)" продублировано.
Таблица 14 - Структура поля "Сертификация лаборатории" (тип данных - LabCertType)
Поле | Тип | Допустимое | Примечание |
Сертификация лаборатории (LabCertification) | string | См. таблицу 15 | Повторяется число раз, равное числу сертификаций |
Таблица 15 - Допустимые значения поля "Сертификация лаборатории"
Значение |
"No validation" (Валидации отсутствуют) |
"ISO/IEC 17025 certification" (Сертификация по ИСО/МЭК 17025) |
"GLP validation" (Валидация в рамках GLP |
"ААВВ certification" (Сертификация ААВВ |
"ISO/ILAC Guidance |
"Unknown" (Неизвестно) |
"Unspecified" (Не определено) |
|
6.4.3.1.6 Поле "Область аккредитации"
Поле "Область аккредитации" должно определять область сертификации лаборатории, которая обрабатывает ДНК. Поле "Область аккредитации" является составным. Структура поля "Область аккредитации" представлена в таблице 16. Допустимые значения поля "Область аккредитации" представлены в таблице 17. Допускается указывать более одной области сертификации.
Таблица 16 - Структура поля "Область аккредитации" (тип данных - SOAType)
Поле | Тип | Допустимое | Примечание |
Область аккредитации (ScopeOfAccreditation) | string | См. таблицу 17 | Повторяется число раз, равное числу аккредитаций |
Таблица 17 - Допустимые значения поля "Область аккредитации"
Значение |
"Nuclear" (Ядерная ДНК) |
"Mitochondrial" (Митохондриальная ДНК) |
"Database" (Базы данных ДНК) |
"Other" (Другое) |
"Unspecified" (Не определено) |
Допускается указывать в поле "Область аккредитации" более одного значения, чтобы представить полный сертификационный статус лаборатории.
6.4.3.1.7 Поле "Тип запроса"
________________
В ИСО/МЭК 19794-14:2013 допущена опечатка - указано поле "Request" вместо поля "Request type".
Поле "Тип запроса" предназначено для определения правовых ограничений и правил. Поле должно иметь тип данных string, допустимые значения представлены в таблицах 18-19.
Таблица 18 - Поле "Тип запроса"
Поле | Тип данных | Допустимое | Примечание |
Значение типа запроса (RequestValue) | RequestValue | - | См. таблицу 17 |
Пользовательские данные (UserDefined) | UserDefinedType | - | См. таблицу 44 |
Таблица 19 - Допустимые значения поля "Значение типа запроса"
Значение |
"DataSubmission" (Предоставление данных) |
"DataSubmissionAndSearch" (Предоставление и поиск данных) |
"Search" (Поиск) |
"UserDefined" (Пользовательские данные) |
6.4.3.1.8 Поле "Результат"
Результат сравнения обозначает, что неидентифицированный или ссылочный тип ДНК передается в базы данных других сторон для поиска совпадений и/или сравнения. Если обнаружены совпадения данного неидентифицированного или ссылочного типа ДНК с соответствующими ссылочными или неидентифицированными типами ДНК в базах данных других сторон, то такие совпадения называются результатом сравнения. Результат может быть записан, только если в поле "Направление связи" записано значение "А" (Answer, ответ). Поле "Результат" должно иметь тип данных string. Поле "Результат" является составным. Структура поля "Результат" представлена в таблице 20.
Таблица 20 - Структура поля "Результат"
Поле | Тип данных | Допустимое | Примечание |
Значение результата (ResuitValue) | ResuitValue | - | См. таблицу 21 |
Пользовательские данные ответа (HitUserDefined) | UserDefinedType | - | См. таблицу 44 |
Пользовательские данные (UserDefined) | UserDefinedType | - | См. таблицу 44 |
Таблица 21 - Допустимые значения поля "Значение результата"
Значение |
"UnableToProcess" (Обработка невозможна) |
"NoHit" (Нет ответа) |
"HitUserDefined" (Пользовательские данные ответа) |
"UserDefined" (Пользовательские данные) |
6.4.3.1.9 Поле "Сообщение об ошибке"
В поле "Сообщение об ошибке" должно быть записано сообщение об ошибке, указывающей на несовместимый тип ДНК, ошибку поиска совпадений и/или связи. Поле должно иметь тип данных string.
6.4.3.1.10 Поле "Дополнительное сообщение"
Поле "Дополнительное сообщение" должно содержать строку с дополнительной информацией или комментариями с типом данных string.
6.4.3.2 Блок "ДНК-типирование" (DNA typing)
Структура блока "ДНК-типирование" представлена в таблице 22.
Таблица 22 - Структура блока "ДНК-типирование"
Поле | Тип | Допустимое | Обязательное/необязательное |
КТП-ДНК-профиль (STR DNA profile) | LocusType | - | Обязательное, если значение в поле "Технология типирования образца" равно "STR". |
Y-КТП-ДНК-профиль (Y-STR DNA profile) | LocusType | - | Обязательное, если значение в поле "Технология типирования образца" равно "Y-STR". |
Данные митохондриальной ДНК (Mitocondrial DNA data) | mtDNAType | - | Обязательное, если значение в поле "Технология типирования образца" равно "mtDNA". |
Данные электрофореграм- мы (Electropherogram data) | EPGType | - | Обязательное, если значение в поле "Технология типирования образца" равно "Electropherogram". |
Пользовательские данные (User defined) | Vendor- | - | Необязательное |
6.4.3.2.1 Поле "КТП-ДНК-профиль"
Поле "КТП-ДНК-профиль" является составным, должно иметь тип данных LocusType. Тип данных LocusType включает поле "Информация о локусе". Структура поля "КТП-ДНК-профиль" представлена в таблице 23.
Таблица 23 - Структура поля "КТП-ДНК-профиль" (тип данных - LocusType)
Поле | Тип данных | Допустимое | Примечание |
Информация о локусе (Locus information) | LocusInfoType | - | Повторяется число раз, равное числу локусов. См. таблицу 24 |
Поле "Информация о локусе" является составным, должно иметь тип данных LocusInfoType и состоять из полей "Заголовок локуса" (Locus header) и "Идентификация аллели" (Allele call). Структура поля "Информация о локусе" представлена в таблице 24.
Таблица 24 - Структура поля "Информация о локусе" (тип данных - LocusInfoType)
Поле | Тип данных | Допустимое | Примечание |
Заголовок локуса | LocusHeaderType | - | См. таблицу 25 |
Идентификация аллели | AlleleCallType | - | Повторяется число раз, равное числу идентификаций аллелей. См. таблицу 26 |
Поле "Заголовок локуса" является составным, должно иметь тип данных LocusHeaderType и состоять из полей "Название маркера локуса" (Name of locus marker) и "Статус" (Status). Структура поля "Заголовок локуса" представлена в таблице 25.
Таблица 25 - Структура поля "Заголовок локуса" (тип данных - LocusHeaderType)
Поле | Тип | Допустимое | Примечание |
Название маркерного локуса | string | - | См. приложение D |
Статус | string | "Normal" (Нормальный), "SilentAllele" (Пустая аллель), "NotDetermined" (Не определено), "NotAnalysed" (Не анализировано) | См. описание ниже |
Статус "Normal" (Нормальный) обозначает, что какие-либо проблемы отсутствуют.
Статус "SilentAllele" (Пустая аллель) обозначает, что аллель не обнаружена.
Статус "NotDetermined" (Не определено) обозначает, что не удалось точно идентифицировать аллель.
Статус "NotAnalysed" (Не анализировано) обозначает, что локус не был анализирован.
Поле "Идентификация аллели" является составным, должно иметь тип данных AlleleCallType и состоять из поля "Оператор" (Operator) и полей "Число идентификации аллели" (Allele call number). Структура поля "Идентификация аллели" представлена в таблице 26.
Таблица 26 - Структура поля "Идентификация аллели" (тип данных - AlleleCallType)
Поле | Тип | Допустимое | Примечание |
Оператор | string | "Equal" (Равно), "LowerLimit" (Нижний предел), "UpperLimit" (Верхний предел), "Range" (Диапазон) | - |
Число 1 идентификации аллели (Allele call number #1) | float | - | Присутствует всегда |
Число 2 идентификации аллели (Allele call number #2) | float | - | Только если в поле "Оператор" указано значение "Range" |
Названия маркеров локусов приведены в приложении D.
6.4.3.2.2 Поле "Y-КТП-ДНК-профиль"
Каждая идентификация локуса Y-КТП должна быть представлена типом LocusType. Названия маркеров локусов приведены в приложении D.
6.4.3.2.3 Поле "Данные митохондриальной ДНК"
Несмотря на полностью завершенное секвенирование мтДНК в качестве инструмента анализа, существуют определенные различия в его интерпретации. Для устранения различий типирования мткДНК (различий от референсного образца), этап интерпретации исключается путем разделения контрольной области на 2 участка (даже при наличии HV3), чтобы обеспечить включение любых вставок / делеций / С-участков. Это позволяет любому получателю использовать данные обычным для него способом (использование или интерпретация полной последовательности методом получателя). Итоговые данные будут полностью совместимы с базой данных получателя с возможностью их обработки.
Контрольная область 1 мтДНК: включает HV1, начиная с 16024 и заканчивая 16569. Длина строки должна быть 546 символов.
Контрольная область 2 мтДНК: включает HV2 и HV3, начиная с 1 и заканчивая 576. Длина строки должна быть 576 символов.
Поле "Данные митохондриальной ДНК" является составным. Структура поля "Данные митохондриальной ДНК" представлена в таблице 27.
Таблица 27 - Структура поля "Данные митохондриальной ДНК" (тип данных - mtDNAType)
Поле | Тип данных | Примечание |
Контрольная область 1 мтДНК (Mito control region 1) | string | См. таблицу 28 |
Контрольная область 2 мтДНК (Mito control region 2) | string | См. таблицу 28 |
Качество 1 мтДНК (Mito DNA Quality 1) | string | - |
Качество 2 мтДНК (Mito DNA Quality 2) | string | - |
В таблице 28 приведены значения символов, установленные IUPAC для исследований типов нуклеотидов ДНК. Допустимый алфавит для последовательности включает символы "А", "Т", "С", "G" и многозначные коды IUPAC. Символ "U не представлен в таблице 28, но часто используется для обозначения химического расщепления "С". Любой участок, который не имеет значения, должен быть установлен в нуль (0).
________________
ИЮПАК - Международный союз теоретической и прикладной химии (IUPAC, International Union of Pure and Applied Chemistry).
Таблица 28 - Значения символов, установленные IUPAC
Значение IUPAC | Определение |
G | Гуанин (Guanine) |
A | Аденин (Adenine) |
T | Тимин (Thymine) |
C | Цитозин (Cytosine) |
R | G, A |
Y | Т, С |
М | А, С |
К | G, Т |
S | G, С |
W | А, Т |
Н | А, С, Т |
В | G, Т, С |
V | G, С, А |
D | G, А, Т |
N | G, А, Т, С |
- | Делеция |
6.4.3.2.4 Поле "Данные электрофореграммы"
Электрофореграмма является изображением результатов анализа, выполняемого при помощи электрофореза при автоматическом секвенировании. Электрофореграммы могут использоваться для получения результатов при генеалогическом ДНК-тестировании, установлении отцовства, секвенировании ДНК, генетической дактилоскопии.
Поле "Данные электрофореграммы" является составным. Структура поля "Данные электрофореграммы" представлена в таблице 29.
Таблица 29 - Структура поля "Данные электрофореграммы" (тип данных - ElectropherogramType)
Поле | Тип данных | Допустимое | Примечание |
Данные электрофореграммы | EPGType | - | См. таблицу 30 |
Данные референсной электрофореграммы (Reference electropherogram data) | EPGRefType | - | См. таблицу 41 |
Данные электрофореграммы митохондриальной последовательности (Electropherogram data for mitochondrial sequence) | EPGmitoType | - | См. таблицу 42 |
Для электрофореграммы должны быть включены следующие необработанные данные (таблица 30):
- данные времени и интенсивности флуоресценции (time and fluorescence strength data);
- данные времени и соответствующих значений пар оснований (time and base pair correspondence data);
- данные панели (panel data);
- бинарные данные (bin data).
Таблица 30 - Структура поля "Данные электрофореграммы" (тип данных - EPGType)
Поле | Тип данных | Допустимое | Примечание |
Данные времени и интенсивности флуоресценции | TFSDType | - | См. таблицу 31 |
Данные времени и соответствующих значений пар оснований | ТВРСТуре | - | См. таблицу 34 |
Данные панели | PanelType | - | См. таблицу 36 |
Бинарные данные | BinType | - | См. таблицу 38 |
Информация о волне локуса включает данные полей "Данные времени и интенсивности флуоресценции" и "Данные времени и соответствующих значений пар оснований".
В поле "Данные времени и интенсивности флуоресценции" содержится значение высоты пика обнаружения флуоресценции и времени (таблицы 31-33). Поле "Данные времени и интенсивности флуоресценции" является составным. Структура поля "Данные времени и интенсивности флуоресценции" представлена в таблице 31.
Таблица 31 - Структура поля "Данные времени и интенсивности флуоресценции" (тип данных - TFSDType)
Поле | Тип данных | Допустимое | Примечание |
Название дорожки (Run name) | string | - | - |
Наименование файла образца (Sample file name) | string | - | - |
Данные красителей электрофореграммы (Electropherogram dye data) | Electropherogram- | - | См. таблицу 32 |
Данные времени 1 электрофореграммы (Electropherogram time data 1) | Electropherogram- | - | Повторяется число раз, равное числу временных данных. См. таблицу 33 |
Таблица 32 - Структура поля "Данные красителей флуоресценции" (тип данных - ElectropherogramDyeDataТуре)
Поле | Тип данных | Допустимое | Примечание |
Наименование красителя 1 (Dye name 1) | string | - | Наименование цвета красителя. |
Таблица 33 - Структура поля "Данные времени электрофореграммы" (тип данных - ElectropherogramTimeDataType)
Поле | Тип данных | Допустимое | Примечание |
Время на дорожке (Time in the run) | integer | - | - |
Интенсивность флуоресценции для красителя 1 (Fluorescence strength for dye 1) | float | - | Повторяется число раз, равное числу красителей |
Значения поля "Данные времени и соответствующих значений пар оснований", как правило, производятся во время электрофоретического анализа. Так как с помощью электрофореза можно обнаружить только время и интенсивность пика, используются маркеры длин. С использованием электрофореграммы маркеров длин (которую можно рассматривать как "референсный образец") рассчитывается соответствие между временем и парами оснований. Значения поля "Данные времени и соответствующих значений пар оснований" являются результатом этого расчета, который содержит время и соответствующее значение пар оснований (таблицы 34-35).
Таблица 34 - Структура поля "Данные времени и соответствующих значений пар оснований" (тип данных - ТВРСТуре)
Поле | Тип данных | Допустимое | Примечание |
Название дорожки | string | - | - |
Наименование файла образца | string | - | - |
Данные соответствия 1 (Correspondence data 1) | Correspondence- | - | Повторяется число |
Таблица 35 - Структура поля "Данные соответствия" (тип данных - CorrespondenceDataType)
Поле | Тип данных | Допустимое | Примечание |
Время на дорожке | integer | - | - |
Длина пар оснований (Base pair size) | float | - | - |
Информация о проверке локусов включает данные полей "Данные панели" и "Бинарные данные".
Поля "Данные панели" и "Бинарные данные" используются для определения идентификации аллелей по электрофореграмме. Поле "Данные панели" определяет набор определений групп для одного или более локусов (таблицы 36-37). Эти данные включают цвет красителя и диапазон корректных размеров ампликона. Данные также включают диапазон высоты электрофоретического пика, в котором аллель должна считаться идентифицированной. В поле "Бинарные данные" указывается диапазон размеров ампликона. Определяется каждая идентификация аллели (число повторов).
Таблица 36 - Структура поля "Данные панели" (тип данных - PanelType)
Поле | Тип данных | Допустимое | Примечание |
Наименование набора праймеров (Primer set name) | string | - | - |
Данные аллели 1 панели (Panel allele data 1) | EPanelAllele- | - | Повторяется число раз, равное числу аллелей. См. таблицу 37 |
Таблица 37 - Структура поля "Данные аллели панели" (тип данных - PanelAlleleDataType)
Поле | Тип | Допустимое | Примечание |
Название локуса (Locus name) | string | - | Название локуса, например, "D8S1179" |
Название красителя (Dye name) | string | - | Краситель, используемый для анализа данного локуса |
Минимальный размер аллели (Minimal allele size) | float | - | - |
Максимальный размер аллели (Maximum allele size) | float | - | - |
Коэффициент шума (Noise ratio) | float | 0.1-1.0 | Отображает, какая интенсивность флуоресценции принимается как достоверная идентификация аллели |
В поле "Бинарные данные" указывается диапазон размеров ампликона (таблицы 38-40). Определяется каждая идентификация аллели (число повторов).
Таблица 38 - Структура поля "Бинарные данные" (тип данных - BinType)
Поле | Тип данных | Допустимое | Примечание |
Наименование набора праймеров | string | - | - |
Бинарные данные локуса 1 (Bin locus data 1) | BinLocus- | - | Повторяется число раз, равное числу бинарных данных локуса. См. таблицу 39 |
Таблица 39 - Структура поля "Бинарные данные локуса" (тип данных - BinLocusDataType)
Поле | Тип данных | Допустимое | Примечание |
Название локуса | string | - | Название локуса, например, "D8S1179" |
Бинарные данные идентификации 1 (Bin call data 1) | BinCallData- | - | Повторяется число раз, равное числу бинарных данных идентификации. См. таблицу 40 |
Таблица 40 - Структура поля "Бинарные данные идентификации" (тип данных - BinCallDataType)
Поле | Тип данных | Допустимое | Примечание |
Идентификация аллели | AlleleCallType | - | См. таблицу 26 |
Среднее значение длины пар оснований (Average base pair size) | float | - | Среднее значение длины пар оснований для данной идентификации |
Отрицательное отклонение длины пар оснований (Minus deviation base pair size) | float | - | Максимальное отрицательное отклонение от среднего значения длины пар оснований |
Положительное отклонение длины пар оснований (Plus deviation base pair size) | float | - | Максимальное положительное отклонение от среднего значения длины пар оснований |
Данные референсной электрофореграммы должны быть указаны в поле "Данные времени и интенсивности флуоресценции" в виде необработанных данных времени и интенсивности флуоресценции (таблица 41).
Таблица 41 - Структура поля "Данные референсной электрофореграммы" (тип данных - EPGRefType)
Поле | Тип данных | Допустимое | Примечание |
Данные времени и интенсивности флуоресценции (Time and fluorescence strength data) | TFSDType | - | См. таблицу 31 |
Электрофоретические данные митохондриальной последовательности должны быть определены в полях "Данные времени и интенсивности флуоресценции" и "Назначение красителя для нуклеотида" (Dye assignment for base) (таблица 42). Поле "Назначение красителя для нуклеотида" должно определять соответствие цвета красителя и типа нуклеотида и, таким образом, содержать два поля "Название красителя" и "Тип нуклеотида" (таблица 43).
Таблица 42 - Структура поля "Электрофоретические данные митохондриальной последовательности" (тип данных - EPGmitoType)
Поле | Тип данных | Допустимое | Примечание |
Данные времени и интенсивности флуоресценции (Time and fluorescence strength data) | TFSD-Type | - | См. таблицу 31 |
Назначение красителя для нуклеотида 1 (Dye assignment for base 1) | Dye-Base- | - | Повторяется число раз, равное числу типов нуклеотидов IUPAC. См. таблицу 43 |
Таблица 43 - Структура поля "Назначение красителя для нуклеотида" (тип данных - DyeBaseAssignType)
Поле | Тип данных | Допустимое | Примечание |
Название красителя | string | - | - |
Тип нуклеотида | string | Значение IUPAC | См. таблицу 27 |
6.4.3.2.5 Поле "Пользовательские данные ДНК" (User defined DNA data)
Структура поля "Пользовательские данные ДНК" представлена в таблице 44.
Таблица 44 - Структура поля "Пользовательские данные ДНК"
Поле | Тип данных | Допустимое значение | Примечание |
Код типа (Type code) | string | - | - |
Данные (Data) | base64Binary | - | Кодировка Base 64 |
Приложение А
(обязательное)
Методология испытаний на соответствие
А.1 Общие положения
Настоящий стандарт определяет формат обмена биометрическими данными для хранения, записи и передачи одного или нескольких представлений ДНК. Каждое представление ДНК сопровождается метаданными, содержащимися в заголовке записи. Настоящее приложение определяет порядок проведения испытаний для проверки корректности записи.
Цель настоящего стандарта не может быть в полной мере достигнута до тех пор, пока биометрические продукты не пройдут испытания на соответствие требованиям настоящего стандарта. Соответствие реализации требованиям является необходимым условием для достижения совместимости между реализациями, поэтому существует необходимость в стандартизированной методологии испытаний на соответствие, тестовых утверждениях и методиках испытаний применительно к конкретным биометрическим модальностям, рассмотренным в настоящем стандарте. Тестовыми утверждениями проверяются наиболее важные требования настоящего стандарта, и соответствие результатов, полученных с помощью комплектов для проведения испытаний на соответствие, будет показывать степень соответствия реализаций настоящему стандарту. Это является причиной разработки данной методологии испытаний на соответствие.
Настоящее приложение является обязательным и предназначено для определения элементов методологии испытаний на соответствие, тестовых утверждений и методик испытаний применительно к настоящему стандарту. Для текущей версии настоящего стандарта содержание настоящего приложения будет доступно в виде отдельного документа (Изменения), дополняющего настоящий стандарт.
Приложение В
(обязательное)
XML-схема данных ДНК
Настоящее приложение содержит пример XML-схемы данных ДНК, который адаптирован и синхронизирован в соответствии с правилами и определениями ИСО/МЭК 19794-1:2011 (Изм.2) (Структура XML-кодирования).
<?xml version="1.0" encoding="UTF-8"?>
<xs:schema xmlns:xs="https://www.w3.org/2001/XMLSchema" xmlns="//standards.iso.org/iso-iec/19794/-14/ed-1" xmlns:dna="//standards.iso.org/iso-iec/19794/-14/ed-1" xmlns:cmn="//standards.iso.org/iso-iec/19794/-l/ed-2/amd/2" targetName- space="//standards.iso.org/iso-iec/19794/-14/ed-1" elementFormDefault="qualified" attributeFormDefault="unqualified">
<xs:annotation>
<xs:documentation>
Настоящим, любому лицу предоставляется бессрочное разрешение на бесплатное использование, копирование, изменение, объединение, опубликование и распространение копий схемы для разработки, внедрения, установки и использования программного обеспечения, разработанного с применением данной схемы при соблюдении следующих условий: схема предоставляется на условиях "как есть", без какой-либо гарантии, явной или подразумеваемой, включая все без исключения подразумеваемые гарантии товарности или пригодности для какой-либо определенной цели. Ни при каких обстоятельствах авторы и владельцы авторского права не несут ответственности за какие-либо претензии, убытки и другие обязательства, возникшие вследствие выполнения обязательств по договору, неосторожности или гражданского правонарушения или других причин, возникших в результате или вследствие использования, или функционирования данной схемы.
</xs:documentation>
</xs:annotation>
<xs:annotation>
<xs:documentation>Данная XML-схема содержит все типы определений (сложные и простые),
которые используются при обмене данных ДHK</xs:documentation>
<xs:documentation>Cтaтyc: v1.0</xs:documentation>
</xs:annotation>
<xs:import namespace="//standards.iso.org/iso-iec/19794/-1/ed-2/amd/2"
schemaLocation="19794-1_common-final.xsd"/>
<xs:simpleType name="LocusStatusType">
<xs:restriction base="xs:string">
<xs:enumeration value="Normal"/>
<xs:enumeration value="SilentAllele"/>
<xs:enumeration value="Undetermined"/>
<xs:enumeration value="NotAnalyzed"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="LocusHeaderType">
<xs:sequence>
<xs:element name="NameOfLocusMarker" type="xs:string"/>
<xs:element name="Status" type="LocusStatusType"/>
</xs:sequence>
</xs:complexType>
<xs:simpleType name="OperatorType">
<xs:restriction base="xs:string">
<xs:enumeration value="Equal"/>
<xs:enumeration value="LowerLimit"/>
<xs:enumeration value="UpperLimit"/>
<xs:enumeration value="Range"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="AlleleCallType">
<xs:sequence>
<xs:element name="Operator" type="OperatorType"/>
<xs:element name="AlleleCallNumber1" type="xs:float"/>
<xs:element name="AlleleCallNumber2" type="xs:float"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="LocusInfoType">
<xs:sequence>
<xs:element name="LocusHeader" type="LocusHeaderType"/>
<xs:element name="AlleleCall" type="AlleleCallType"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="LocusType">
<xs:sequence>
<xs:element name="LocusInformation" type="LocusInfoType" minOccurs="O"/>
</xs:sequence>
</xs:complexType>
<xs:simpleType name="IupacType">
<xs:restriction base="xs:string">
<xs:pattern value="[GATCRYMKSWHBVDN]+"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="MtDnaType">
<xs:sequence>
<xs:element name="MitoControlRegion1" type="IupacType"/>
<xs:element name="MitoControlRegion2" type="IupacType"/>
<xs:element name="MitoDnaQuality1" type="xs:string"/>
<xs:element name="MitoDnaQuality2" type="xs:string"/>
</xs:sequence>
</xs:complexType>
<xs: complexType name="UserDefinedType">
<xs:sequence>
<xs:element name="TypeCode" type="xs:string" minOccurs="O"/>
<xs:element name="Data" type="xs:base64Binary"/>
</xs:sequence>
</xs:complexType>
</xs:complexType>
<xs:complexType name="ElectropherogramTimeDataType">
<xs:sequence>
<xs:element name="TimeIntheRun" type="xs:integer"/>
<xs:element name="FluorescenceStrengths" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="FluorescenceStrength" type="xs: float"
minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="ElectropherogramDyeDataType">
<xs:sequence>
<xs:element name="DyeName" type="xs:string" minOccurs="O"
maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="TfsdType">
<xs:sequence>
<xs:element name="RunName" type="xs:string"/>
<xs:element name="SampleFileName" type="xs:string"/>
<xs:element name="ElectropherogramDyeDataSet" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="ElectropherogramDyeData" type="ElectropherogramDyeDataType" minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
<xs:element name="ElectropherogramTimeDataSet" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="ElectropherogramTimeData" type="ElectropherogramTimeDataType" minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="CorrespondenceDataType">
<xs:sequence>
<xs:element name="TimeInTheRun" type="xs:integer"/>
<xs:element name="BasePairSize" type="xs:float" minOccurs="O"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="TbpcType">
<xs:sequence>
<xs:element name="RunName" type="xs:string"/>
<xs:element name="SampleFileName" type="xs:string"/>
<xs:element name="CorrespondenceDataSet" minOccurs="0">
<xs:complexType>
<xs:sequence>
<xs:element name="CorrespondenceData" type="CorrespondenceDataType" minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="PanelAlleleDataType">
<xs:sequence>
<xs:element name="LocusName" type="xs:string"/>
<xs:element name="DyeName" type="xs:string" minOccurs="O"/>
<xs:element name="MinAlleleSize" type="xs: float" minOccurs="0"/>
<xs:element name="MaxAlleleSize" type="xs: float" minOccurs="0"/>
<xs:element name="NoiseRatio" type="xs:float" minOccurs="O"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="PanelType">
<xs:sequence>
<xs:element name="PrimerSetName" type="xs:string"/>
<xs:element name="PanelAlleleDataSet" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="PanelAlleleData" type="PanelAlleleDataType"
minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="BinCallDataType">
<xs:sequence>
<xs:element name="AlleleCall" type="AlleleCallType"/>
<xs:element name="AverageBasePairSize" type="xs:float"/>
<xs:element name="MinusDeviationBasePairSize" type="xs:float"/>
<xs:element name="PlusDeviationBasePairSize" type="xs:float"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="BinLocusDataType">
<xs:sequence>
<xs:element name="LocusName" type="xs:string"/>
<xs:element name="BinCallDataSet" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="BinCallData" type="BinCallDataType" minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="BinType">
<xs:sequence>
<xs:element name="PrimerSetName" type="xs:string"/>
<xs:element name="BinLocusDataSet" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="BinLocusData" type="BinLocusDataType" minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="EpgType">
<xs:sequence>
<xs:element name="TfsdData" type="TfsdType"/>
<xs:element name="TbpcData" type="TbpcType"/>
<xs:element name="PanelData" type="PanelType"/>
<xs:element name="BinData" type="BinType"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="EpgRefType">
<xs:sequence>
<xs:element name="TfsdData" type="TfsdType"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="DyeBaseAssignType">
<xs:sequence>
<xs:element name="DyeName" type="xs:string"/>
<xs:element name="BaseType" type="IupacType"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="EpgMitoType">
<xs:sequence>
<xs:element name="TfsdData" type="TfsdType"/>
<xs:element name="DyeBaseAssignment" type="DyeBaseAssignType" minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="ElectropherogramType">
<xs:sequence>
<xs:element name="SampleData" type="EpgType"/>
<xs:element name="ReferenceData" type="EpgRefType"/>
<xs:element name="MitoEpgData" type="EpgMitoType"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="DnaTypingType">
<xs:sequence>
<xs:element name="StrDnaType" type="LocusType" minOccurs="O"/>
<xs:element name="YstrDnaType" type="LocusType" minOccurs="O"/>
<xs:element name="MtDnaData" type="MtDnaType" minOccurs="O"/>
<xs:element name="Electropherogram" type="ElectropherogramType"
minOccurs="O"/>
<xs:element name="UserDefinedDnaData" type="UserDefinedType"
minOccurs="O"/>
</xs:sequence>
</xs:complexType>
<xs:simpleType name="RequestValue">
<xs:restriction base="xs:string">
<xs:enumeration value="DataSubmission"/>
<xs:enumeration value="DataSubmissionAndSearch"/>
<xs:enumeration value="Search"/>
<xs:enumeration value="UserDefined"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="ResultValue">
<xs:restriction base="xs:string">
<xs:enumeration value="UnableToProcess"/>
<xs:enumeration value="NoHit"/>
<xs:enumeration value="HitUserDefined"/>
<xs:enumeration value="UserDefined"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="RequestType">
<xs:sequence>
<xs:element name="RequestValue" type="RequestValue"/>
<xs:element name="UserDefined" type="UserDefinedType" minOccurs="0"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="ResultType">
<xs:sequence>
<xs:element name="ResultValue" type="ResultValue"/>
<xs:element name="HitUserDefined" type="UserDefinedType" minOccurs="0"/>
<xs:element name="UserDefined" type="UserDefinedType" minOccurs="0"/>
</xs:sequence>
</xs:complexType>
<xs:simpleType name="LabCertificationType">
<xs:restriction base="xs:string">
<xs:enumeration value="NoValidation"/>
<xs:enumeration value="IsoIec17025Certification"/>
<xs:enumeration value="GlpValidation"/>
<xs:enumeration value="AabbCertification"/>
<xs:enumeration value="IsoILacGuild19Accreditation"/>
<xs:enumeration value="Unknown"/>
<xs:enumeration value="Unspecified"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="SoaType">
<xs:restriction base="xs:string">
<xs:enumeration value="Nuclear"/>
<xs:enumeration value="Mitochondrial"/>
<xs:enumeration value="Database"/>
<xs:enumeration value="Other"/>
<xs:enumeration value="Unspecified"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="DnaTypingDataType">
<xs:sequence>
<xs:element name="DateAndTimeOfAnalaysis" type="xs:dateTime"/>
<xs:element name="BatchId" type="xs:string" minOccurs="O"/>
<xs:element name="DnaProfileId" type="xs:string" minOccurs="1"/>
<xs:element name="KitId" type="xs:string" minOccurs="O"/>
<xs:element name="LabCertifications" minOccurs="0">
<xs:complexType>
<xs:sequence>
<xs: element name="LabCertification" type="LabCertificationType"
minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
<xs:element name="ScopeOfAccreditations" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="ScopeOfAccreditation" type="SoaType"
minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
<xs:element name="Request" type="RequestType"/>
<xs:element name="Result" type="ResultType"/>
<xs:element name="ErrorMessage" type="xs:string" minOccurs="O"/>
<xs:element name="SupplementaryMessage" type="xs:string"
minOccurs="0"/>
</xs:sequence>
</xs:complexType>
<xs:simpleType name="SampleCategoryType">
<xs:restriction base="xs:string">
<xs:enumeration value="Arrestee"/>
<xs:enumeration value="ClaimedBiologicalChild"/>
<xs:enumeration value="ClaimedBiologicalFather"/>
<xs:enumeration value="ClaimedBiologicalMother"/>
<xs:enumeration value="ClaimedBiologicalSibling"/>
<xs:enumeration value="ClaimedBiologicalSpouse"/>
<xs:enumeration value="ActualBiologicalChild"/>
<xs:enumeration value="ActualBiologicalFather"/>
<xs:enumeration value="ActualBiologicalMother"/>
<xs:enumeration value="ActualBiologicalSibling"/>
<xs:enumeration value="ActualBiologicalSpouse"/>
<xs:enumeration value="AdoptiveBiologicalChild"/>
<xs:enumeration value="AdoptiveBiologicalFather"/>
<xs:enumeration value="AdoptiveBiologicalMother"/>
<xs:enumeration value="AdoptiveBiologicalSibling"/>
<xs:enumeration value="AdoptiveBiologicalSpouse"/>
<xs:enumeration value="ConvictedOffender"/>
<xs:enumeration value="UnknownForensic"/>
<xs:enumeration value="Insurgent"/>
<xs:enumeration value="KnownSuspectedTerrorist"/>
<xs:enumeration value="MaternalRelative"/>
<xs:enumeration value="MissingPerson"/>
<xs:enumeration value="PaternalRelative"/>
<xs:enumeration value="KnownSuspect"/>
<xs:enumeration value="UnidentifiedLiving"/>
<xs:enumeration value="UnidentifiedDead"/>
<xs:enumeration value="KnownVictim"/>
<xs:enumeration value="Detainee"/>
<xs:enumeration value="Other"/>
<xs:enumeration value="Unspecified"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="SampleCellularType">
<xs:restriction base="xs:string">
<xs:enumeration value="Blood"/>
<xs:enumeration value="Bone"/>
<xs:enumeration value="BuccalCell"/>
<xs:enumeration value="CommingledBiologicalMaterial"/>
<xs:enumeration value="Hair"/>
<xs:enumeration value="Saliva"/>
<xs:enumeration value="Semen"/>
<xs:enumeration value="Skin"/>
<xs:enumeration value="SweatFingerprint"/>
<xs:enumeration value="Tissue"/>
<xs:enumeration value="ToothPulp"/>
<xs:enumeration value="Other"/>
<xs:enumeration value="Unknown"/>
<xs:enumeration value="Unspecified"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="SampleTypingTechnologyType">
<xs:restriction base="xs:string">
<xs:enumeration value="Str"/>
<xs:enumeration value="YStr"/>
<xs:enumeration value="MtDna"/>
<xs:enumeration value="Electropherogram"/>
<xs:enumeration value="UserDefinedTyping"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="SpecimenContributorType">
<xs:restriction base="xs:string">
<xs:maxLength value="7"/>
<xs:enumeration value="Known"/>
<xs:enumeration value="Unknown"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="GeoLocationType">
<xs:sequence>
<xs:element name="Latitude" type="xs:float"/>
<xs:element name="Longitude" type="xs:float"/>
</xs:sequence>
</xs:complexType>
<xs:simpleType name="SpecimenIDType">
<xs:restriction base="xs:string">
<xs:maxLength value="24"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="PedigreeMbrStatusType">
<xs:restriction base="xs:string">
<xs:enumeration value="Known"/>
<xs:enumeration value="Unknown"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="GenderType">
<xs:restriction base="xs:string">
<xs:enumeration value="Male"/>
<xs:enumeration value="Female"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="PedigreeMbrType">
<xs:sequence>
<xs:element name="PedigreeMemberId" type="xs:integer"/>
<xs:element name="SpecimenId" type="SpecimenIDType"/>
<xs:element name="MotherId" type="xs:integer"/>
<xs:element name="FatherId" type="xs:integer"/>
<xs:element name="PedigreeMemberStatus" type="PedigreeMbrStatusType"/>
<xs:element name="Gender" type="GenderType"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="PedigreeType">
<xs:sequence>
<xs:element name="PedigreeId" type="xs:integer"/>
<xs:element name="PedigreeMembers" minOccurs="0">
<xs:complexType>
<xs:sequence>
<xs:element name="PedigreeMember" type="PedigreeMbrType" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="PedigreeTreeType">
<xs:sequence>
<xs:element name="Pedigree" type="PedigreeType" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="RepresentationHeaderType">
<xs:sequence>
<xs:element name="SampleCollectionDate" type="xs:dateTime"/>
<xs:element name="SampleCategory" type="SampleCategoryType"/>
<xs:element name="SampleCellularType" type="SampleCellularType"/>
<xs:element name="SampleTypingTechnology"
type="SampleTypingTechnologyType"/>
<xs:element name="SpecimenContributor"
type="SpecimenContributorType"/>
<xs:element name="SampleCollectionMethod" type="xs:string" minOccurs="O"/>
<xs:element name="SampleCollectionLocation" type="xs:string" minOccurs="O"/>
<xs:element name="SampleCollectionGeoLocation" type="GeoLocationType" minOccurs="O"/>
<xs:element name="PedigreeTrees" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="PedigreeTree" type="PedigreeTreeType" minOccurs="0" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
<xs:complexType name="DnaRepresentationType">
<xs:sequence>
<xs:element name="RepresentationHeader" type="RepresentationHeaderType"/>
<xs:element name="DnaTypingData" type="DnaTypingDataType"/>
<xs:element name="DnaTyping" type="DnaTypingType"/>
</xs:sequence>
</xs:complexType>
<xs:simpleType name="FormatIdentifierType">
<xs:restriction base="xs:string">
<xs:maxLength value="3"/>
<xs:enumeration value="Dna"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="CommunicationDirectionType">
<xs:restriction base="xs:string">
<xs:enumeration value="Request"/>
<xs:enumeration value="Answer"/>
</xs:restriction>
</xs:simpleType>
<xs:simpleType name="EntityType">
<xs:restriction base="xs:string">
<xs:enumeration value="G"/>
<xs:enumeration value="GM"/>
<xs:enumeration value="GR"/>
<xs:enumeration value="I"/>
<xs:enumeration value="IM"/>
<xs:enumeration value="IR"/>
<xs:enumeration value="O"/>
<xs:enumeration value="OM"/>
<xs:enumeration value="OR"/>
<xs:enumeration value="U"/>
<xs:enumeration value="UM"/>
<xs:enumeration value="UR"/>
</xs:restriction>
</xs:simpleType>
<xs:complexType name="PartyType">
<xs:sequence>
<xs:element name="NationalityCode" type="xs:string"/>
<xs:element name="EntityName" type="xs:string"/>
<xs:element name="PersonName" type="xs:string"/>
</xs:sequence>
</xs:complexType>
<xs:complexType name="GeneralHeaderType">
<xs:sequence>
<xs:element name="FormatIdentifier" type="FormatIdentifierType"/>
<xs:element name="Version" type="cmn:VersionType"/>
<xs:element name="CommunicationDirection" type="CommunicationDirectionType"/>
<xs:element name="SendingParty" type="PartyType"/>
<xs:element name="ReceivingParty" type="PartyType"/>
<xs:element name="EntityType" type="EntityType"/>
<xs:element name="DateAndTimeOfDataProcessing" type="xs:dateTime"/>
</xs:sequence>
</xs:complexType>
<xs:element name="Dna">
<xs:complexType>
<xs:sequence>
<xs:element name="GeneralHeader" type="GeneralHeaderType"/>
<xs:element name="Representations" minOccurs="O">
<xs:complexType>
<xs:sequence>
<xs:element name="Representation"type="DnaRepresentationType" maxOccurs="unbounded"/>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:sequence>
</xs:complexType>
</xs:element>
</xs:schema>
Приложение С
(обязательное)
Идентификаторы наборов реагентов
С.1 Идентификаторы наборов реагентов
Таблица С.1 - Идентификаторы наборов реагентов
Идентификатор набора реагентов (название набора реагентов для ПЦР | Изготовитель | Описание набора реагентов |
________________
| ||
Profiler | Applied | AmpFISTR® Profiler® (Артикул 403038) |
Profiler Plus | Applied | AmpFISTR® Profiler Plus® (Артикул 4303326) |
COfiler | Applied | AmpFISTR® COfiler® (Артикул 4305246) |
Profiler Plus and COfiler | Applied | AmpFISTR® Profiler Plus® and COfiler® (Артикул 4305979) |
Profiler Plus ID | Applied | AmpFISTR® Profiler Plus® ID (Артикул 4330284) |
Profiler Plus ID and COfiler | Applied | AmpFISTR® Profiler Plus® ID and COfiler® (Артикул 4330621) |
SGM Plus | Applied | AmpFISTR® SGM Plus® (Артикул 4307133) |
Identifier | Applied | AmpFISTR® Identifiler® (Артикул 4322288) |
Identifier Direct | Applied | AmpFISTR® Identifiler® Direct PCR Amplification Kit (Артикул 4408580) |
Identifier Plus | Applied | AmpFISTR® Identifiler® Plus PCR Amplification Kit (Артикул 4427368) |
MiniFiler | Applied | AmpFISTR® MiniFiler |
NGM | Applied | AmpFISTR® NGM |
NGM SEIect | Applied | AmpFISTR® NGM SElect |
SEfiler Plus | Applied | AmpFISTR® SEfiler Plus |
Yfiler | Promega | AmpFISTR® Yfiler® PCR Amplification Kit (Артикул 4359513) |
PowerPlex 1.1 | Promega | PowerPlex® 1.1 (Номера по каталогу DC6091 /6090) |
PowerPlex 1.2 | Promega | PowerPlex® 1.2 (Номера по каталогу DC6100 /6101) |
PowerPlex 1.1 и PowerPlex 1.2 | Promega | PowerPlex® 1.1 и PowerPlex® 1.2 (Номера по каталогу DC6501 / DC6500) |
________________ | ||
PowerPlex 2.1 | Promega | PowerPlex® 2.1 (Номера по каталогу DC 6471 /6470) |
PowerPlex 16 | Promega | PowerPlex® 16 (Номера по каталогу DC 6531 /6530) |
PowerPlex 16 BIO | Promega | PowerPlex® 16 ВIO (Номера по каталогу DC 6541 /6540) |
PowerPlex 16 HS | Promega | PowerPlex® 16 HS System (Номера по каталогу DC2101 / DC2100) |
PowerPlex 18D | Promega | PowerPlex® 18D System (Номера по каталогу DC 1802 /1808) |
PowerPlex CS7 | Promega | PowerPlex® CS7 System, Custom (Номер по каталогу Х6613) |
PowerPlex ES | Promega | PowerPlex® ES System (Номера по каталогу DC6731 / DC6730) |
PowerPlex ESX 16 | Promega | PowerPlex® ESX 16 System (Номера по каталогу DC6711 / DC6710) |
PowerPlex ESI 16 | Promega | PowerPlex® ESI 16 System (Номера по каталогу DC6771 / DC6770) |
PowerPlex ESX 17 | Promega | PowerPlex® ESX 17 System (Номера по каталогу DC6721 / DC6720) |
PowerPlex ESI 17 | Promega | PowerPlex® ESI 17 System (Номера по каталогу DC6781 / DC6780) |
PowerPlex ESX 16 и PowerPlex ESI 16 | Promega | PowerPlex® ESX 16 и PowerPlex® ESI 16 Systems Bundle (Номера по каталогу DC6792 / DC6793) |
PowerPlex ESX 17 и PowerPlex ESI 17 | Promega | PowerPlex® ESX 17 and PowerPlex® ESI 17 Systems Bundle (Номера по каталогу DC6790 / DC6791) |
PowerPlex S5 | Promega | PowerPlex® S5 System (Номера по каталогу DC6951 / DC6950) |
PowerPlex Y | Promega | PowerPlex® Y System (Номера по каталогу DC6761 / DC6760) |
Monoplex D5S818 | Promega | PowerPlex® 16 и ES Monoplex System, D5S818 (Номер по каталогу DC6601) |
Monoplex D7S820 | Promega | PowerPlex® 16 и ES Monoplex System, D7S820 (Номер по каталогу DC6621) |
Monoplex D13S317 | Promega | PowerPlex® 16 и ES Monoplex System, D13S317 (Номер no каталогу DC6611) |
Monoplex D16S539 | Promega | PowerPlex® 16 и ES Monoplex System, D16S539 (Номер no каталогу DC6631) |
Monoplex TH01 | Promega | PowerPlex® 16 и ES Monoplex System, TH01 (Номер по каталогу DC6561) |
Monoplex TPOX | Promega | PowerPlex® 16 и ES Monoplex System, TPOX (Номер по каталогу DC6681) |
Monoplex CSF1PO | Promega | PowerPlex® 16 и ES Monoplex System, CSF1PO (Номер no каталогу DC6641) |
Monoplex vWA | Promega | PowerPlex® 16 и ES Monoplex System, vWA (Номер по каталогу DC6661) |
Monoplex Penta E | Promega | PowerPlex® 16 и ES Monoplex System, Penta E (Номер no каталогу DC6591) |
Monoplex Penta D | Promega | PowerPlex® 16 и ES Monoplex System, Penta D (Номер по каталогу DC6651) |
Monoplex SE33 | Promega | PowerPlex® 16 и ES Monoplex System, SE33 (Номер по каталогу DC6751) |
Monoplex D3S1358 | Promega | PowerPlex® 16 и ES Monoplex System, D3S1358 (Номер no каталогу DC6551) |
Monoplex D21S11 | Promega | PowerPlex® 16 и ES Monoplex System, D21S11 (Номер по каталогу DC6571) |
Monoplex D18S51 | Promega | PowerPlex® 16 и ES Monoplex System, D18S51 (Номер по каталогу DC6581) |
Monoplex D8S1179 | Promega | PowerPlex® 16 и ES Monoplex System, D8S1179 (Номер no каталогу DC6671) |
Monoplex FGA | Promega | PowerPlex® 16 и ES Monoplex System, FGA (Номер по каталогу DC6691) |
GammaSTR® Multiplex | Promega | GenePrint® GammaSTR® Multiplex D16S539, D7S820, D13S317, D5S818 (Номера по каталогу DC6071 / DC6070) |
CSF1PO, TPOX, TH01, vWA | Promega | GenePrint® CSF1PO, TPOX, TH01, vWA Multiplex (Номера no каталогу DC6301 / DC6300) |
F13A01, FESFPS, F13B, LPL | Promega | GenePrint® F13A01, FESFPS, F13B, LPL Multiplex (Номера no каталогу DC6311 / DC6310) |
Monoplex D5S818 | Promega | Monoplex D5S818 (Номер по каталогу DC6161) * недоступен для покупки |
Monoplex D7S820 | Promega | Monoplex D7S820 (Номер по каталогу DC6141) * недоступен для покупки |
Monoplex D13S317 | Promega | Monoplex D13S317 (Номер по каталогу DC6151)* недоступен для покупки |
Monoplex D16S539 | Promega | Monoplex D16S539 (Номер по каталогу DC6131) * недоступен для покупки |
Monoplex TH01 | Promega | Monoplex ТН01 (Номер по каталогу DC5081) * недоступен для покупки |
Monoplex TPOX | Promega | Monoplex TPOX (Номер по каталогу DC5111) * недоступен для покупки |
Monoplex CSF1PO | Promega | Monoplex CSF1РО (Номер по каталогу DC5091) * недоступен для покупки |
Monoplex vWA | Promega | Monoplex vWA (Номер по каталогу DC5141) * недоступен для покупки |
Investigator ESSple | Qiagen | Investigator ESSplex Kit (Номера по каталогу 381515/381517) |
Investigator ESSplex SE | Investigator ESSplex SE Kit (Номера по каталогу 381525 / 381527 | |
Investigator Decaplex SE | Investigator Decaplex SE Kit (Номера по каталогу 381025 / 381027) | |
Investigator IDplex | Investigator IDplex Kit (Номера по каталогу 381615 / 381617) | |
Investigator Nonaplex ESS | Investigator Nonaplex ESS Kit (Номера по каталогу 381315 / 381317) | |
Investigator Hexaplex ESS | Investigator Hexaplex ESS Kit (Номера по каталогу 380615 / 380617) | |
Investigator Triplex DSF | Investigator Triplex DSF Kit (Номера по каталогу 380325 / 380327) | |
Investigator Triplex AFS QS | Investigator Triplex AFS QS Kit (Номера по каталогу 380315 / 380317) | |
Investigator HDplex | Investigator HDplex Kit (Номера по каталогу 381213/381215) | |
Investigator Argus X-12 | Investigator Argus X-12 Kit (Номера по каталогу 383213 / 383215) | |
Investigator Argus Y-12 QS | Investigator Argus Y-12 QS Kit (Номера по каталогу 383615 / 383617) | |
С.2 Библиография
Для получения соответствующей информации может быть использован следующий официальный сайт в сети Интернет:
//www.nist.gov/itl/iad/ig/ansi_standard.cfm
Приложение D
(обязательное)
Локусы ДНК
D.1 Локусы ДНК
Таблица D.1- Локусы ДНК
ИЛД | Аутосомный локус КТП | ИЛД | Локус Х-КТП | ИЛД | Локус Y-КТП |
________________ | |||||
1 | Амелогенин | 1 | DXS10011 | 1 | DXYS156 |
2 | CD4 | 2 | DXS10066 | 2 | DYF371 |
3 | CSF1PO | 3 | DXS10067 | 3 | DYF385a |
4 | D10S1248 | 4 | DXS10068 | 4 | DYF385b |
5 | D10S1435 | 5 | DXS10069 | 5 | DYF395 |
6 | D10S2325 | 6 | DXS10074 | 6 | DYF397a |
7 | D11S4463 | 7 | DXS10075 | 7 | DYF397b |
8 | D12ATA63 | 8 | DXS10076 | 8 | DYF397c |
9 | D12S391 | 9 | DXS10077 | 9 | DYF397d |
10 | D13S317 | 10 | DXS10078 | 10 | DYF399a |
11 | D14S1434 | 11 | DXS10079 | 11 | DYF399b |
12 | D16S539 | 12 | DXS101 | 12 | DYF399c |
13 | D17S1301 | 13 | DXS10101 | 13 | DYF406S1 |
14 | D17S974 | 14 | DXS10103 | 14 | DYF408a |
15 | D18S51 | 15 | DXS10129 | 15 | DYF408b |
16 | D18S853 | 16 | DXS10130 | 16 | DYF408c |
17 | D19S433 | 17 | DXS10131 | 17 | DYF408d |
18 | D1GATA113 | 18 | DXS10132 | 18 | DYF411a |
19 | D1S1627 | 19 | DXS10133 | 19 | DYF411b |
20 | D1S1656 | 20 | DXS10134 | 20 | DYS19 (=DYS394) |
21 | D1S1677 | 21 | DXS10135 | 21 | DYS385a |
22 | D20S1082 | 22 | DXS10146 | 22 | DYS385b |
23 | D20S482 | 23 | DXS10147 | 23 | DYS388 |
24 | D21S11 | 24 | DXS10148 | 24 | DYS389I |
25 | D21S2055 | 25 | DXS10159 | 25 | DYS389II |
26 | D22S1045 | 26 | DXS10160 | 26 | DYS390 |
27 | D2S1338 | 27 | DXS10161 | 27 | DYS391 |
28 | D2S1360 | 28 | DXS10162 | 28 | DYS392 |
29 | D2S1776 | 29 | DXS10163 | 29 | DYS393 |
30 | D2S441 | 30 | DXS10164 | 30 | DYS395S1a |
31 | D3S1358 | 31 | DXS10165 | 31 | DYS395S1b |
32 | D3S1545 | 32 | DXS6789 | 32 | DYS413a |
33 | D3S1744 | 33 | DXS6795 | 33 | DYS413b |
34 | D3S3053 | 34 | DXS6797 | 34 | DYS425 |
35 | D3S4529 | 35 | DXS6799 | 35 | DYS426 |
36 | D4S2364 | 36 | DXS6800 | 36 | DYS434 |
37 | D4S2366 | 37 | DXS6801 | 37 | DYS435 |
38 | D4S2408 | 38 | DXS6803 | 38 | DYS436 |
39 | D5S2500 | 39 | DXS6804 | 39 | DYS437 |
40 | D5S818 | 40 | DXS6807 | 40 | DYS438 |
41 | D6S1017 | 41 | DXS6809 | 41 | DYS439 |
42 | D6S1043 | 42 | DXS6810 | 42 | DYS441 |
43 | D6S474 | 43 | DXS7130 | 43 | DYS442 |
44 | D7S1517 | 44 | DXS7132 | 44 | DYS444 |
45 | D7S820 | 45 | DXS7133 | 45 | DYS445 |
46 | D8S1115 | 46 | DXS7423 | 46 | DYS446 |
47 | D8S1132 | 47 | DXS7424 | 47 | DYS447 |
48 | D8S1179 | 48 | DXS8377 | 48 | DYS448 |
49 | D9S1122 | 49 | DXS8378 | 49 | DYS449 |
50 | D9S2157 | 50 | DXS981 | 50 | DYS450 |
51 | F13A01 | 51 | DXS9895 | 51 | DYS452 |
52 | F13B | 52 | DXS9898 | 52 | DYS454 |
53 | FESFPS | 53 | DXS9902 | 53 | DYS455 |
54 | FGA | 54 | DXS9905 | 54 | DYS456 |
55 | GABA | 55 | DXS9906 | 55 | DYS458 |
56 | LPL | 56 | DXS9907 | 56 | DYS459a |
57 | Penta_B | 57 | DXS9908 | 57 | DYS459b |
58 | Penta_C | 58 | DXYS156 | 58 | DYS460 |
59 | Penta_D | 59 | GATA144D04 | 59 | DYS461 |
60 | Penta_E | 60 | GATA165B12 | 60 | DYS462 |
61 | SE33 | 61 | GATA172D05 | 61 | DYS463 |
62 | TH01 | 62 | GATA31E08 | 62 | DYS464a |
63 | TPOX | 63 | HPRTB | 63 | DYS464b |
64 | vWA | 64 | HUMARA | 64 | DYS464c |
65 | DYS464d | ||||
66 | DYS464e | ||||
67 | DYS464f | ||||
68 | DYS464g | ||||
69 | DYS472 | ||||
70 | DYS481 | ||||
71 | DYS485 | ||||
72 | DYS487 | ||||
73 | DYS490 | ||||
74 | DYS492 | ||||
75 | DYS495 | ||||
76 | DYS511 | ||||
77 | DYS520 | ||||
78 | DYS522 | ||||
79 | DYS527a (=DYF401a) | ||||
80 | DYS527b (=DYF401b) | ||||
81 | DYS531 | ||||
82 | DYS532 | ||||
83 | DYS534 | ||||
84 | DYS537 | ||||
85 | DYS557 | ||||
86 | DYS565 | ||||
87 | DYS568 | ||||
88 | DYS570 | ||||
89 | DYS572 | ||||
90 | DYS576 | ||||
91 | DYS578 | ||||
92 | DYS590 | ||||
93 | DYS594 | ||||
94 | DYS607 | ||||
95 | DYS617 | ||||
96 | DYS635 (Y-GATA-C4) | ||||
97 | DYS640 | ||||
98 | DYS641 | ||||
99 | DYS643 | ||||
100 | DYS650 | ||||
101 | DYS652 | ||||
102 | DYS709 | ||||
103 | DYS710 | ||||
104 | DYS712 | ||||
105 | DYS714 | ||||
106 | DYS715 | ||||
107 | DYS716 | ||||
108 | DYS717 | ||||
109 | DYS724a (=CDYa) | ||||
110 | DYS724b (=CDYb) | ||||
111 | DYS725a | ||||
112 | DYS725b | ||||
113 | DYS725c | ||||
114 | DYS725d | ||||
115 | DYS726 | ||||
116 | YCAIIa | ||||
117 | YCAIIb | ||||
118 | Y-GATA-A10 | ||||
119 | Y-GATA-H4 | ||||
120 | Y-GGAAT-1B07 | ||||
D.2 Библиография
Для получения соответствующей информации может быть использован следующий официальный сайт в сети Интернет:
//www.nist.gov/itl/iad/ig/ansi_standard.cfm
Приложение ДА
(справочное)
Сведения о соответствии ссылочных международных стандартов национальным стандартам
Таблица ДА.1
Обозначение ссылочного международного стандарта | Степень | Обозначение и наименование соответствующего национального стандарта |
ISO/IEC 19794-1:2011 | IDT | ГОСТ ISO/IEC 19794-1-2015 "Информационные технологии. Биометрия. Форматы обмена биометрическими данными. Часть 1. Структура" |
ISO/IEC 19794-1:2011 (Изм.2) | - | * |
* Соответствующий национальный стандарт отсутствует. Примечание - В настоящей таблице использовано следующее условное обозначение степени соответствия стандарта: - IDТ - идентичный стандарт. | ||
УДК 004.93'1:006.89:006.354 | ОКС 35.040 | П85 | IDТ |
Ключевые слова: информационные технологии, биометрия, форматы обмена биометрическими данными, данные ДНК, ДНК, мтДНК, ДНК-профиль | |||
Электронный текст документа
и сверен по:
, 2019