ГОСТ Р 58505-2019/ISO/TS 20428:2017
НАЦИОНАЛЬНЫЙ СТАНДАРТ РОССИЙСКОЙ ФЕДЕРАЦИИ
ИНФОРМАТИЗАЦИЯ ЗДОРОВЬЯ
Элементы данных и их метаданные для описания структурированной информации о клиническом геномном секвенировании в электронных медицинских картах
Health informatics. Data elements and their metadata for describing structured clinical genomic sequence information in electronic health records
ОКС 35.240.80
Дата введения 2020-05-01
Предисловие
1 ПОДГОТОВЛЕН Федеральным государственным унитарным предприятием "Российский научно-технический центр информации по стандартизации, метрологии и оценке соответствия" (ФГУП "") на основе собственного перевода на русский язык англоязычной версии документа, указанного в пункте 4
2 ВНЕСЕН Техническим комитетом по стандартизации ТК 468 "Информатизация здоровья"
3 УТВЕРЖДЕН И ВВЕДЕН В ДЕЙСТВИЕ Приказом Федерального агентства по техническому регулированию и метрологии от 29 августа 2019 г. N 575-ст
4 Настоящий стандарт идентичен международному документу ISO/TS 20428:2017* "Информатизация здоровья. Элементы данных и их метаданные для описания структурированной информации о клиническом геномном секвенировании в электронных медицинских картах" (ISO/TS 20428:2017 "Health informatics - Data elements and their metadata for describing structured clinical genomic sequence information in electronic health records", IDT)
________________
* Доступ к международным и зарубежным документам, упомянутым в тексте, можно получить, обратившись в Службу поддержки пользователей. - .
5 ВВЕДЕН ВПЕРВЫЕ
Правила применения настоящего стандарта установлены в статье 26 Федерального закона от 29 июня 2015 г. N 162-ФЗ "О стандартизации в Российской Федерации". Информация об изменениях к настоящему стандарту публикуется в ежегодном (по состоянию на 1 января текущего года) информационном указателе "Национальные стандарты", а официальный текст изменений и поправок - в ежемесячном информационном указателе "Национальные стандарты". В случае пересмотра (замены) или отмены настоящего стандарта соответствующее уведомление будет опубликовано в ближайшем выпуске ежемесячного информационного указателя "Национальные стандарты". Соответствующая информация, уведомление и тексты размещаются также в информационной системе общего пользования - на официальном сайте Федерального агентства по техническому регулированию и метрологии в сети Интернет (www.gost.ru)
Предисловие к ISO/TS 20428
Международная организация по стандартизации (ИСО) является Всемирной федерацией национальных органов по стандартизации (комитеты - члены ИСО). Работу по подготовке международных стандартов, как правило, осуществляют технические комитеты ИСО. Каждый комитет - член ИСО, проявляющий интерес к тематике, по которой учрежден технический комитет, имеет право быть представленным в этом комитете. Международные организации, государственные и негосударственные, связанные с деятельностью ИСО, также принимают участие в работе. ИСО тесно сотрудничает с Международной электротехнической комиссией (МЭК) по всем вопросам стандартизации в области электротехники.
Процедуры, используемые для разработки настоящего стандарта, и процедуры, предусмотренные для его дальнейшего ведения, описаны в Директивах ИСО/МЭК, часть 1. В частности, следует отметить различные критерии утверждения, требуемые для разных типов документов ИСО. Проект настоящего стандарта разработан в соответствии с редакционными правилами Директив ИСО/МЭК, часть 2, по адресу: www.iso.org/directives.
Ряд элементов настоящего стандарта могут быть предметом патентных прав. Международная организация ИСО не должна нести ответственность, частично или полностью, за идентификацию таких прав. Сведения о патентных правах, идентифицированных при разработке настоящего стандарта, будут указаны во введении и/или в перечне полученных ИСО объявлениях о патентном праве (www.iso.org/patents).
Любое торговое название, использованное в настоящем стандарте, является информацией, предоставляемой для удобства, а не свидетельством в пользу того или иного товара либо той или иной компании.
Для пояснения значений конкретных терминов и выражений ИСО, относящихся к оценке соответствия, а также для получения информации о соблюдении Международной организацией ИСО принципов ВТО по техническим барьерам в торговле (ТБТ) можно воспользоваться унифицированным указателем ресурса (URL): Foreword - Supplementary information (www.iso.org/iso/foreword.html).
Ответственность за содержание настоящего стандарта несет Технический комитет ISO/TC 215 "Информатизация здоровья".
Введение
На фоне быстрого совершенствования технологий секвенирования на первый план выдвигается клиническое секвенирование как одна из методик выполнения задач персонализированной медицины и прецизионной медицины. В открытом доступе имеется масса данных секвенирования с клинической информацией [1]. Кроме того, в медицинской практике получает широкое распространение полногеномное клиническое секвенирование [2]. Многие стационарные медицинские учреждения приступили к секвенированию полного генома, полного экзома пациентов или таргетных генов с использованием технологий секвенирования нового поколения. Такие геномные данные, полученные посредством технологий секвенирования нового поколения, можно использовать как для клинико-диагностических целей, так и для выбора надлежащих медицинских препаратов, в том числе научные исследования. Вопросам управления геномными и клиническими данными придают особое значение в прецизионной медицине, клинических испытаниях и прикладных исследованиях [3].
Однако до сих пор не существует международного стандарта для представления результатов клинического секвенирования со структурным форматом электронного медицинского архива, в связи с чем данные геномных тестов, необходимые врачам-клиницистам, поступают некачественно оформленными, что снижает эффективность работы специалистов. Имеется несколько соответствующих стандартов по моделированию результатов генетических исследований (например, ИСО 25720 и несколько документов HL7 рабочей группы клинической геномики HL7). Однако указанные стандарты или проекты по большей части сфокусированы на результатах традиционных генетических исследований, рассчитанных на анализ одного гена. На основании быстрого совершенствования и принятия технологий секвенирования нового поколения, позволяющих выявлять разнообразные генетические варианты на геномном уровне, можно говорить о том, что разработка стандарта по представлению данных секвенирования таким образом, чтобы они стали полезными для клиницистов [4], - по-прежнему актуальная задача.
Для включения структурированного отчета о клиническом секвенировании в электронный медицинский архив следует определить все необходимые поля данных, а также метаданные по каждому выбранному полю, например: какой вокабулярий (словарь), в частности дескрипции генов и/или коды заболеваний, можно заносить в отдельные поля. В ISO/TS 215 предложен язык GSVML (Genomic Sequence Variation Markup Language - язык разметки вариаций геномной последовательности) для интероперабельности геномных вариантов, особенно для данных однонуклеотидного полиморфизма ОНП (SNP) [5]. В организации HL7 (Health Level Seven International, //www.hl7.org/) также работают над созданием модели анализа области геномики с использованием версии 3 HL7 [6] и спецификации FHIR (Fast Healthcare Interoperability Resources - резистентных ресурсов интероперабельности здравоохранения) [7]. Для удобства применения геномной информации разработаны ресурсы SMART (Substitutable Medical Applications, Reusable Technologies - взаимозаменяемые медицинские приложения, технологии повторного использования) и FHIR Genomics [8], [9]. Консорциум стандартов обмена клиническими данными CDISC (Clinical Data Interchange Standard Consortium) опубликовал справочник применения табличных данных в фармакогеномике и генетике (Study Tabulation Model Implementation Guide: Pharmacogenomics/Genetics) [10]. Несколько международных организаций, например Глобальный альянс для геномики и здравоохранения GA4GH (Global Alliance for Genomics and Health), Действующий геномный консорциум (Actionable Genome Consortium), а также Группа представления и интеграции генетической информации через EHR (DIGITIzE, Displaying and Integrating Genetic Information Through the EHR) Института медицины в США (Institute of Medicine in US), пытались разработать аналогичные стандарты. Рабочая группа Комиссии по обеспечению качества Лаборатории Американского колледжа медицинской генетики и геномики (American College of Medical Genetics and Genomics Laboratory Quality Assurance Committee) опубликовала стандарты по клинической лаборатории ACMG для секвенирования нового поколения [11]. Кроме того, высвободился доступный через сеть инструментарий, соединяющий генотипическую информацию с фенотипической информацией, при этом очень полезным может оказаться обмен информацией и ее использование в персонализированной медицине [12].
В настоящем стандарте для возможности использования геномных данных пациента на основе клинического секвенирования для решения терапевтических задач, а также для клинических испытаний и исследований могут быть разработаны метаданные для отчета о клиническом секвенировании для занесения в электронный медицинский архив. Далее дается разъяснение, каким образом и где именно могут быть занесены в вышеназванные поля конкретные соответствующие терминологические системы, описывающие геномы и/или заболевания. Посредством определения необходимых полей со структурным форматом по закодированным данным с соблюдением терминологических принципов, например презентация и управление концепцией, настоящий стандарт может быть полезным при внедрении поддержки принятия клинических решений.
1 Область применения
Настоящий стандарт определяет элементы данных и необходимые для них метаданные для выполнения структурированного отчета о клиническом геномном секвенировании для занесения в электронный медицинский архив с упором на геномные данные, созданные посредством технологии секвенирования нового поколения.
Настоящий стандарт:
- определяет построение структурированного отчета о клиническом геномном секвенировании (см. раздел 5);
- определяет поля необходимых данных и их метаданных для структурированного отчета о клиническом геномном секвенировании (см. раздел 6);
- определяет дополнительные данные (см. раздел 7);
- охватывает изменчивость на уровне ДНК по образцам, взятым у человека, с использованием полногеномного секвенирования, полноэкзомного секвенирования и таргетного секвенирования (таргетные генетические панели, направленные на конкретное заболевание) при помощи технологий секвенирования нового поколения. Несмотря на то что транскриптомное секвенирование и иные технологии важны для обеспечения лучшего ухода за пациентом и позволяют проведение прецизионной медицины, настоящий стандарт касается исключительно изменчивости на уровне ДНК;
- охватывает в основном применение в медицинской практике и клинические исследования, например клинические испытания и прикладные исследования с использованием клинических данных с учетом, в частности, деидентификации или согласия пациента. Фундаментальные исследования и иные научные направления не включены в объем настоящего стандарта;
- не охватывает другие биологические виды, например геномы вирусов и микробов, а также
- не охватывает методики секвенирования по Сенгеру.
2 Нормативные ссылки
Нормативные ссылки в настоящем стандарте отсутствуют.
3 Термины и определения
В настоящем стандарте применены следующие термины с соответствующими определениями.
ИСО и МЭК поддерживают терминологические базы данных для использования в стандартизации по следующим адресам:
- интерактивная платформа для просмотра ИСО - //www.iso.org/obp
- электропедия МЭК - //www.electropedia.org
3.1
аллель (allele): Одна из нескольких альтернативных форм гена, которая возникает на одном и том же локусе гомологичных хромосом, отделяется при редукционном делении и может рекомбинироваться после слияния гамет. [ИСО 16577:2016, пункт 3.6] |
3.2 доброкачественный (benign): Генетический вариант с убедительными доказательствами против его патогенности.
3.3 биоматериал (biomaterial): Материалы, взятые из тела человека, например ткань, кровь, плазма или моча.
3.4
хромосома (chromosome): Структура, содержащая дискретные участки ДНК и белки и несущая генетическую информацию, которая конденсируется во время деления ядер и образует характерным образом сформированные тельца. [ИСО 19238:2014, пункт 2.7] |
3.5 клиническое секвенирование (clinical sequencing): Технологии секвенирования нового или более позднего поколения с использованием образцов, взятых у человека для проведения клинической практики и клинических испытаний.
3.6 ClinVar: Находящийся в открытом доступе архив отчетов о взаимосвязях между генетическими вариантами ДНК человека и фенотипом с подтверждающими данными.
Примечание 1 - //www.ncbi.nlm.nih.gov/clinvar/.
3.7 вариант числа копий; ВЧК (copy number variation; CNV): Вариант числа копий одного участка ДНК или более.
3.8 каталог соматических мутаций в раковых опухолях (Catalogue of Somatic Mutations in Cancer); COSMIC: Интерактивная база данных о соматических мутациях в раковых клетках.
Примечание 1 - //cancer.sanger.ac.uk/cosmic
3.9 dbSNP: База данных SNP (ОНП, однонуклеотидных полиморфизмов), предоставляемая Национальным центром биотехнологий (NCBI).
Примечание 1 - https://www.ncbi.nlm.nih.gov/SNP/
3.10 делеция (deletion): Мутация, в процессе которой часть хромосомы или последовательности ДНК теряется во время репликации ДНК.
3.11
дезоксирибонуклеиновая кислота; ДНК (deoxyribonucleic acid; DNA): Молекула, кодирующая генетическую информацию в ядрах клеток. [ИСО 25720:2009, пункт 4.7] |
3.12
секвенирование ДНК (DNA sequencing): Определение последовательности нуклеотидных оснований (аденин, гуанин, цитозин и тимин) в молекуле ДНК. Примечание 1 - Секвенирование, как правило, описывается с 5'-конца. [ISO/TS 17822-1:2014, пункт 3.20] |
3.13 экзом (exome): Часть генома, сформированная экзонами.
3.14
ген (gene): Базовый элемент наследственного материала, кодирующий белок или субъединицу белка и контролирующий его экспрессию. [ИСО 11238:2012, пункт 2.1.16] |
3.15 генетическая панель (gene panel): Методика секвенирования целевых (таргетных) генов в геноме.
3.16 геномная медицина (genomic medicine): Медицинская дисциплина, включающая использование геномной информации об индивидууме как части его клинического лечения (например, для диагностики или принятия терапевтических решений), а также эффекты лечения и выводы исследования по его клиническому использованию.
3.17 генеративная линия (germline): Ряд зародышевых клеток, каждая из которых произошла или развилась из более ранних клеток в этом ряду, рассматриваемая как продолжение последовательных генераций организма.
3.18 вставка-делеция (indel): Вставка (3.19) или/и делеция (3.10).
3.19 вставка (insertion): Добавление одной или нескольких пар нуклеотидных оснований в последовательность ДНК.
3.20 инверсия (inversion): Хромосомная перестройка, при которой сегмент хромосомы меняется из конца в конец.
3.21 большая вставка-делеция (large indel): Вставка или делеция до ~1 кб (килобаз).
3.22 вероятно доброкачественный (likely benign): Генетический вариант с доказательствами против его патогенности.
Примечание 1 - Целевое тестирование членов семьи группы риска не рекомендуется.
3.23 вероятно патогенные (likely pathogenic): Генетический вариант с доказательствами его патогенности.
3.24 патогенный (pathogenic): Генетический вариант с крайне убедительными доказательствами его патогенности.
3.25 пренатальный/фетальный (prenatal/fetal): Образец биоматериала плода, забранный в дородовый период.
3.26 прочтенная последовательность [прочтение] (sequence read [read]): Фрагментированные нуклеотидные последовательности, используемые для реконструкции оригинальной последовательности в технологиях секвенирования нового поколения.
3.27 тип прочтения (read type): Тип прогона в секвенаторе.
Примечание 1 - Может быть односторонним или парным.
Примечание 2 - Одностороннее: при одностороннем чтении секвенатор считывает с одного конца фрагмента до другого конца.
Примечание 3 - Парно (двухстороннее): парное чтение производят с одного конца последовательности до другого конца, затем наступает второй круг чтения с противоположного конца.
3.28 референсная последовательность (reference sequence): Цифровая база данных нуклеотидной последовательности, собранная учеными в качестве стандартного (репрезентативного) образца генома человека.
3.29
рибонуклеиновая кислота; РНК (ribonucleic acid; RNA): Полимер рибонуклеотидов, возникающий в двухцепочной или одноцепочной форме. [ИСО 22174:2005, пункт 3.1.3] |
3.30
вариант последовательности (sequence variation), [вариант последовательности ДНК] (DNA sequence variation), [вариант] (variation): Различия последовательности ДНК у индивидуумов в популяции. Примечание 1 - Вариант подразумевает CNV (3.7), делецию (3.10), вставку (3.19), вставку-делецию (3.18), малую вставку-делецию (3.32), большую вставку-делецию (3.20) и SNP (3.31). [ИСО 25720:2009, пункт 4.8] |
3.31
однонуклеотидный полиморфизм (single nucleotide polymorphism; SNP): Однонуклеотидный вариант в последовательности ДНК, относительно часто встречающийся в популяции. Примечание 1 - Произносится "снип". [ИСО 25720:2009, пункт 4.23] |
3.32 малая вставка-делеция (small indel): Вставка или делеция 2 ~100 нуклеотидов.
3.33 соматическая клетка (somatic cell): Клетки тела в отличие от клеток зародышевой линии.
3.34 биологический образец (biological specimen), [биообразец] (biospecimen), [образец] (specimen): Проба ткани, биологической жидкости, пищи или иной субстанции, которая собирается или приобретается для поддержки определения, диагностики, лечения, смягчения или профилактики заболевания, расстройства или аномального физического состояния либо его симптомов.
3.35
обследуемый (subject of care): Любое лицо, пользующееся медицинской помощью, или его потенциальный пользователь. [ISO/TS 22220:2011, пункт 3.2] |
3.36 захват мишени (target capture): Методика захвата геномных участков изучаемой области из пробы ДНК перед секвенированием.
3.37 неопределенное значение (uncertain significance): Вариант с ограниченными и/или противоречивыми доказательствами в отношении патогенности.
3.38 полноэкзомное секвенирование (whole exome sequencing; WES): Методика секвенирования всех белоккодирующих генов в геноме.
3.39 полногеномное секвенирование (whole genome sequencing; WGS): Методика единовременного определения полной последовательности ДНК генома организма.
4 Сокращения
В настоящем стандарте применены следующие сокращения:
ACMG - Американская коллегия медицинской генетики и геномики (American College of Medical Genetics and Genomics);
COSMIC - каталог соматических мутаций в раковых опухолях (Catalogue of Somatic Mutations in Cancer);
CPIC - Консорциум по внедрению клинической фармакогенетики (Clinical Pharmacogenetics Implementation Consortium);
EBI - Европейский институт биоинформатики (European Bioinformatics Institute);
FHIR - возможности здравоохранения к быстрому взаимодействию (Fast Healthcare Interoperability Resources);
HGNC - Комитет по номенклатуре генов человека в составе HUGO (HUGO Gene Nomenclature Committee);
HGVS - Общество изучения вариативности генома человека (Human Genome Variation Society);
HUGO - Международная организация по изучению генома человека (Human Genome Organization);
IARC - Международное агентство по изучению рака (International Agency for Research on Cancer);
LOINC - Наименования и коды идентификаторов логического наблюдения (Logical Observation Identifiers Names and Codes);
NCBI - Национальный центр биотехнологической информации (National Center for Biotechnology Information);
NCCN - Национальная всеобщая онкологическая сеть (National Comprehensive Cancer Network);
NGS - секвенирование нового поколения (Next Generation Sequencing);
SNP - однонуклеотидный полиморфизм (Single Nucleotide Polymorphism);
SPREC - стандартное преаналитическое кодирование (Standard Preanalytical Code);
WES - полноэкзомное секвенирование (Whole Exome Sequencing);
WGS - полногеномное секвенирование (Whole Genome Sequencing).
5 Сценарий варианта использования
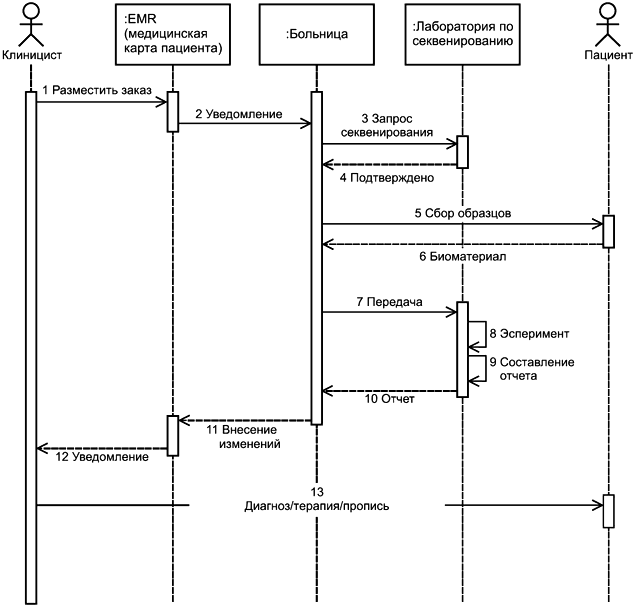
Упрощенный вариант использования для составления отчета по клиническому геномному секвенированию показан на рисунке 1. Вначале клиницист размещает заказ на проведение геномного секвенирования с применением системы электронного медицинского архива (шаг 1 на рисунке 1). После размещения заказа ответственное отделение выполняет запрос на секвенирование ДНК в службу секвенирования (шаг 2). Указанная служба секвенирования может находиться внутри больничного учреждения или представлять собой лабораторию за пределами больничного учреждения (шаг 3). При подтверждении заказа служба секвенирования запрашивает образец от пациента (шаг 4). Больничное отделение берет образец у пациента (шаги 5, 6). Можно также использовать предварительно взятые образцы, например образец из биобанка, образцы, взятые по предыдущим заказам на лабораторные или патологические исследования. Биоматериал пациентов доставляют в лабораторию (шаг 7). После его получения служба секвенирования проводит анализ данных секвенирования (шаг 8) и подготавливает отчет (шаг 9). Указанный отчет направляют в запросившее его лечебное учреждение (шаг 10) и в отчет вносят изменения по электронному медицинскому архиву (шаг 12). В результате направивший заказ клиницист ставит диагноз или проводит соответствующее лечение (шаг 13). Пациент может получить копию заключения.
|
Рисунок 1 - Абстрагированный порядок составления отчета по клиническому геномному секвенированию
Возможные варианты использования для заказов клинического секвенирования доступно изложены в "HL7 VERSION 3 Domain Analysis Model: Clinical Sequencing" [6].
6 Построение отчета о клиническом секвенировании
6.1 Общая часть
Структурированный отчет о клиническом секвенировании в основном состоит из двух частей: резюмирующая часть и детальное содержание, как показано на рисунке 2. Резюмирующая часть может включать в себя ряд подгрупп из необходимых сегментов, для того чтобы оказать помощь клиницистам при быстром просмотре наиболее важных сведений в сжатой форме [13], [14]. Детальное содержание должно включать в себя все необходимые сегменты и избранные дополнительные сегменты.
Настоящий стандарт лишь определяет элементы данных и их метаданных для отчета о структурированном клиническом секвенировании в электронном медицинском архиве. Таким образом, компоновку отчета можно планировать, основываясь на решении лечебного учреждения, если включены все элементы, изложенные в настоящем стандарте.
|
Рисунок 2 - Построение отчета о клиническом секвенировании
6.2 Общая интерпретация в резюмирующей части
В резюмирующей части должна быть представлена общая интерпретация об исследовании генома с кратким описанием: идентифицирован, не идентифицирован, недоказателен или носитель. В таблице 1 приведена краткая информация по каждой интерпретации. "Идентифицирован" свидетельствует об обнаружении варианта, объясняющего состояние пациента. "Не идентифицирован" означает отсутствие вариантов, идентифицированных как правдоподобно уместные для диагностической индикации. "Недоказателен" подразумевает, что не найдено четкое объяснение состояния пациента либо по причине того, что идентифицированы исключительно варианты неизвестного значения, либо по причине того, что идентифицирован только единственный гетерозиготный вариант для рецессивного состояния. "Носитель" представляет собой идентификацию вариантов, выявленных при скрининге на скрытое носительство рецессивной патологии [11].
Таблица 1 - Общая интерпретация в резюмирующей части
Интерпретация | Комментарии |
Идентифицирован | Обнаружение варианта, объясняющего состояние пациента |
Не идентифицирован | Отсутствие варианта, идентифицированного как правдоподобно уместный для диагностической индикации |
Недоказателен | Отсутствие четкого объяснения состояния пациента |
Носитель | Обнаружение вариантов при скрининге на скрытое носительство рецессивной патологии |
6.3 Детальное содержание
Детальное содержание состоит из двух частей, а именно - необходимые и выбранные дополнительные поля. Примером может послужить отчет о генетическом испытании HL7 Implementation Guide for CDA Release 2, однако данный отчет сфокусирован исключительно на тестировании одного гена и не охватывает технологию секвенирования нового поколения.
Обязательные поля главным образом сфокусированы на оказании помощи клиницистам посредством представления необходимой геномной информации, а также результатов интерпретации и соответствующей терапии. Они включают в себя все необходимые поля для клинической практики. Дополнительные поля обеспечивают для клиницистов более детальную информацию, ими можно воспользоваться при проведении прикладных исследований по определенным этапам, например деидентификация или согласие пациента. В [13] поля данных по шаблону отчета о молекулярном генетическом тестировании разделены на категории: "необходимые", "дополнительные", "вероятные" и "необязательные" на основании исследования. Поля данных в настоящем стандарте повторно разделены на категории для выполнения отчета о структурированном клиническом секвенировании посредством рассмотрения имеющегося отчета о секвенировании в произвольном текстовом формате.
7 Поля и их номенклатура обязательных данных
7.1 Общая часть
Выбор обязательных полей производится для клинической практики. Информация, которая может быть описана только в отчете о клиническом секвенировании, включена в обязательные поля для минимизации длины отчета о клиническом секвенировании. Остальная релевантная информация может быть включена в дополнительные поля или другие клинические отчеты в электронный медицинский архив. Краткая информация об элементах данных и их метаданных изложена в таблице 2.
Таблица 2 - Элементы данных и их метаданные для заполнения обязательных полей
Элементы данных | Метаданные (исходные) | ||
Заказы | Код заказа по | Код заказа | LOINC |
по клиническому секвенированию | клиническому секвенированию | Информация о заказе секвенирования | Текст |
Дата и время | Дата заказа | ИСО 8601 | |
Дата взятия образцов | |||
Дата получения заказа | |||
Дата отчета | |||
Дата создания приложения | |||
Информация об образце | ISO/TS 22220:2011 | ||
Информация об | Идентификаторы | ISO/TS 22220:2011 | |
обследуемом | ФИО | ||
Дата рождения | ИСО 8601 | ||
Пол | ISO/TS 22220:2011 | ||
Национальность | HL7 v3 Code System Race | ||
Данные о законном уполномоченном лице - заказчике клинического секвенирования | ISO/TS 27527:2010 | ||
Лаборатория- | Базовая информация | Текст | |
исполнитель | Информация о составителе отчета | Текст | |
Информация о легально верифицированном лице по отчету о секвенировании | ISO/TS 27527:2010 | ||
Сопутствующие заболевания и фенотипы | ICD | ||
Данные | Тип образца | SPREC | |
о биоматериалах | Класс геномного источника в биоматериале | LOINC | |
Состояние образца | Текст | ||
Генетические | Символы и названия генов | HGNC | |
варианты | Информация о | Обозначение | HGVS |
выявленных при | Эффект вариантов | Текст | |
секвенировании вариантах | Идентификатор варианта | Уникальный идентификатор базы данных | |
Классификация вариантов | Патогенность | ENUM ("патогенный", "вероятно патогенный", "неопределенного значения", "вероятно доброкачественный", "доброкачественный") | |
Клиническая значимость | ENUM ("идентифицированный", "вероятно идентифицированный", "неопределенный", "неидентифицированный") | ||
Рекомендуемая | Лекарственное средство | ИСО 11615 | |
терапия | Информация о клиническом испытании | Идентификатор клинического испытания | |
Общепринятые протоколы, относящиеся к варианту | Текст | ||
Другие рекомендации | Текст | ||
| |||
7.2 Заказы на клиническое секвенирование
7.2.1 Общая часть
При оформлении заказа на клиническое геномное секвенирование необходимо присвоить код заказа и осуществить ввод информации по требуемой дате.
7.2.2 Код заказа клинического секвенирования
7.2.2.1 Код заказа
Соответствующие заказы клинического секвенирования должны быть представлены через систему LOINC (наименования и коды идентификаторов логического наблюдения) с использованием ее версии. Коды LOINC по указанным заказам клинического секвенирования в настоящее время отсутствуют, за исключением молекулярно-генетического кода. В то же время можно использовать в качестве альтернативы другие международные, национальные или ведомственные системы кодирования, например CPT (Современная врачебная терминология по процедурам) США имеет в наличии коды для секвенирования.
7.2.2.2 Информация по заказу клинического секвенирования
Если в коде заказа невозможно представить полное описание цели клинического секвенирования, можно подробно описать секвенирование посредством дополнительного произвольного текста, например наименование секвенирования панели с генной информацией.
7.2.3 Дата и время
Для отчета о клиническом секвенировании необходимо в отчет заносить различные форматы даты и времени в зависимости от отставания времени. Все даты и время следует представлять в отчете по ИСО 8601.
7.2.3.1 Дата заказа
Датой заказа является дата заказа клиницистом необходимой информации о клиническом геномном секвенировании.
7.2.3.2 Дата принятия заказа
Датой принятия заказа служит дата получения и подтверждения заказа клинического геномного секвенирования.
7.2.3.3 Дата взятия образца
Дата взятия образца означает дату взятия образца у пациента.
7.2.3.4 Дата составления отчета
Дата составления отчета - это дата составления лабораторией-исполнителем отчета о секвенировании.
7.2.3.5 Дата создания приложения
Дата создания приложения - это дата создания лабораторией-исполнителем приложения о предыдущем отчете с использованием текущей информации. Анализируемые каналы информации или ссылочная база данных обновляются за счет совершенствования технологии. Референсное секвенирование регулярно обновляется. Кроме того, секвенирование подлежит обновлению с точки зрения правовых аспектов. Таким образом, лаборатория-исполнитель должна создавать дополнение к отчету о существующем секвенировании на основании запроса клинициста или правоохранительных органов.
7.2.4 Информация об образце
Информацию об образце можно представлять посредством кода типа идентификатора субъекта в соответствии с ИСО 22220:2011.
Пример 13-S-048435_A1 - Номер патологии: ИСО 22220:2011 (обозначение идентификатора обследуемого): 13-S-048435_A1, идентификатор географического района обследуемого: 1 (локальный), орган, запросивший идентификатор обследуемого: AMC (медицинский центр ABC), 02 (номер специальности - патология).
7.3 Информация об обследуемом
7.3.1 Общая часть
Информация по обследуемому, чей образец подвергся секвенированию, должна быть представлена в соответствии с ИСО 22220:2011.
Пример - 12345678 - ИСО 22220:2011 (обозначение идентификатора обследуемого): 12345678, идентификатор географического района обследуемого: 1 (локальный), орган, запросивший идентификатор обследуемого: AMC (медицинский центр ABC), 01 (уникальный идентификатор запросившего органа).
7.3.2 Идентификаторы обследуемого
Необходимо указать уникальные идентификаторы обследуемых.
7.3.3 ФИО обследуемого
Необходимо указать ФИО обследуемого.
7.3.4 Дата рождения обследуемого
Необходимо указать дату рождения обследуемого, для того чтобы определить возраст пациента. Дату рождения следует установить в соответствии с ИСО 8601.
7.3.5 Пол обследуемого
Пол обследуемого следует указывать в соответствии с ИСО 22220:2011.
7.3.6 Национальность обследуемого
Национальность обследуемого необходимо указывать для представления его/ее генетического происхождения и при помощи системы кодирования HL7 v3 Code System Race (https://www.hl7.org/fhir/v3/Race/index.html). В качестве альтернативы, при наличии национальных стандартов, можно использовать системы кодирования, приведенными в этих стандартах, например Отраслевым руководством US FDA. Collection of Race and Ethnicity Data in Clinical Trials [15].
7.4 Информация по легально уполномоченному лицу, заказывающему клиническое секвенирование
7.4.1 Общая часть
Информацию по легально уполномоченному лицу, заказывающему клиническое секвенирование, можно приводить в соответствии с ISO/TS 26527:2010.
Детальные позиции указанного поля, например ФИО заказывающего терапевта, его/ее медицинскую специальность или номера контрактов, могут быть выбраны лечебными учреждениями-исполнителями или лабораториями.
7.5 Лаборатория-исполнитель
7.5.1 Общая часть
Необходимо указать лабораторию, выполняющую секвенирование.
7.5.2 Основная информация по лаборатории-исполнителе
Необходимо указать лабораторию-исполнитель и контактную информацию, например сообщить телефонные номера и адреса электронной почты. Данная информация должна быть приведена в формате произвольного текста, пока не выпущен соответствующий стандарт.
7.5.3 Информация о составителе отчета
В отчет должна быть включена информация о личности составителя, создавшего отчет о результатах секвенирования. Данные могут быть внесены в соответствии с ISO/TS 26527:2010 или в произвольной форме.
7.5.4 Информация о легально верифицированном лице по отчету о секвенировании
Информация о легально верифицированном терапевте может быть представлена в соответствии с ISO/TS 26527:2010.
7.6 Сопутствующие заболевания и фенотипы
По возможности, в отчет необходимо включать сопутствующие заболевания и фенотипы с использованием кодов ICD или кода SNOMED-CT. Другие фенотипы, не входящие в классификацию по ICD или SNOMED-CT, могут быть указаны с помощью Онтологии человеческого фенотипа (Human Phenotype Ontology) (//human-phenotype-ontology.github.io/).
В силу того что даже одиночный вариант может иметь множество сопутствующих фенотипов, включая заболевания, всевозможные сопутствующие заболевания, необходимо их перечислять, основываясь на уровне достоверности.
В этом поле необходимо указывать только заболевания или фенотипы, сопутствующие найденным фенотипам. Ранее известные заболевания обследуемого, не являющиеся сопутствующими данным вариантам, должны быть исключены из этого поля.
7.7 Информация о биоматериале
7.7.1 Общая часть
Необходимо дать описание информации по образцу, взятому у пациента.
7.7.2 Типы проб
Типы проб можно указывать посредством стандартного ПРЕаналитического кода (SPREC) V2.0 Общества "International Society for Biological and Environmental Repositories" [16].
Пример - BLD (кровь), BUF (лейкоцитное покрытие), CEN (некровеносная ткань), SEM (семя).
7.7.3 Класс геномного источника в биоматериале
Геномный источник образца, взятого у пациента, необходимо разбивать на следующие категории: 1) источник генеративной линии; 2) соматический; 3) пренатальный/эмбриональный; 4) вероятно генеративной линии; 5) вероятно соматический; 6) вероятно пренатальный/эмбриональный или 7) неизвестное геномное происхождение. Данная категоризация основана на данных LOINCS - Список ответов LL378-1.
7.7.4 Состояния образца, которые могут ограничивать достоверность испытаний
Описание специфических состояний, которые могут влиять на достоверность геномного секвенирования или ограничивать его, необходимо представлять в текстовом формате.
Пример - Информация о доброкачественности опухоли должна быть приведена в процентном выражении.
7.8 Генетические варианты
7.8.1 Общая часть
В отчете необходимо перечислять все найденные варианты в соответствии с их релевантностью относительно показаний для проведения тестирования пациента. Однако в обязательном поле должны быть перечислены варианты, имеющие соответствующие формы терапии. Перечисление остальных вариантов должно быть приведено в дополнительных полях.
Как указано в 7.9, варианты можно классифицировать по категориям на основании того, что для найденного варианта имеется надлежащая терапия. При ее отсутствии в клинической практике варианты лечения не представляют смысла. Таким образом, в обязательное поле следует включать только варианты при наличии терапии.
Информация по вариантам должна включать в себя нижеприведенные данные.
7.8.2 Символы и наименования генов
Символ и наименование гена, содержащего создаваемые варианты, должны быть указаны посредством HGNC [Комиссия по номенклатуре (наименованию) генов человеческих в составе HUGO (Международной организации по изучению генома человека)]. HGNC утвердила символы генов и, возможно, использование идентификаторов HGNC. Такие символы нечувствительны к регистру.
Пример - Одобренный HGNC символ: BRCA1, HGNC ID: HGND:1100.
HGNC - это некоммерческий орган, совместно финансируемый Национальным институтом генома человека (NHGRI) и Независимым международным фондом Welcome Trust (Великобритания). Данные организации действуют при поддержке HUGO (Международной организации по изучению генома человека).
7.8.3 Информация о вариативности секвенирования
7.8.3.1 Общая часть
Вариант секвенирования следует приводить на основе терминологии HGVS (Общество по изучению варианта генома человека, //www.hgvs.org/). Для получения более подробной информации можно использовать данные об изменениях аминокислот.
Пример - c.76A>T_p.Asn26Tyr.
7.8.3.2 Терминология HGVS
Описание изменений последовательностей в ДНК подробно разъяснено по адресу //varnomen.hgvs.org/recommendations/DNA/
Пример - Можно представить следующие примеры различных вариантов секвенирования:
- замены: в HGVS ">" означает замену на уровне ДНК (например, замена нуклеотида А на Т в позиции 76: c.76A>T);
- делеции: в HGVS "del" означает делецию (например, c.76delA);
- дупликации: в HGVS "dup" означает дупликацию (например, c.76dupA);
- инсерции: в HGVS "ins" означает инсерцию (например, c.76_77insG);
- инсерции/делеции: в HGVS "delins" означает инсерцию/делецию (c.112_117delinsTG). Инсерции/делеции также описываются как делеция, следующая за инсерцией;
- инверсии: в HGVS ‘inv’ означает инверсию (например, c.76_83inv);
- конверсии: в HGVS ‘con’ означает конверсию. Пример "g.123_678conNG_012232.1:g.9456_10011" дает описание конверсии гена, заменяющей нуклеотиды 123 до 678 в референсной геномной последовательности нуклеотидами 9456 до 10011 из последовательности, как представлено в файле GenBank NG_012232.1;
- транслокации: в HGVS описание транслокаций дано на молекулярном уровне с использованием формата "t(X;4)(p21.2;q35)", последующим обычным нумерованием и указанием точки разрыва локализованной транслокации. t(X;4)(p21.2;q35)(c.857+101_857+102) означает точку разрыва транслокации, локализованной в интроне между кодирующими ДНК нуклеотидами 857+101 и 857+102, соединяя сегменты хромосом Xp21.2 и 4q34.
7.8.3.3 Эффекты вариативности
Разъяснения по типам вариантов можно давать в произвольной форме.
Пример - Замена, делеция, дупликация, инсерция, инверсия и конверсия.
Дополнительно можно приводить в скобках эффекты мутации по белковой функции, например миссенс, нонсенс-мутация и молчащая мутация.
Пример - Замена (миссенс).
7.8.3.4 Идентификатор варианта секвенирования
Все известные варианты можно указывать в отчете, используя их уникальные идентификаторы.
Если вариант секвенирования имеет идентификатор dbSNP ID (//www.ncbi.nlm.nih.gov/projects/SNP/), то этот вариант можно указывать посредством идентификатора dbSNP ID. Префикс "rs" представляет идентификатор dbSNP ID (например, rs10000), который поддерживается NCBI.
ClinVar является базой данных по зависимостям среди вариантов и фенотипов человека (с подтверждающими данными), поддерживаемой NCBI (//www.ncbi.nlm.nih.gov/clinvar/). Если используется идентификатор ClinVar ID, то перед указанным идентификатором необходимо ставить префикс "ClinVar:" (например, ClinVar:17661), как в стандартном профиле для генетики HL7 FHIR.
Если вариант представляет собой соматический вариант, можно использовать идентификатор COSMIC (Каталог соматических мутаций в раковых опухолях) с другими идентификаторами (//cancer.sanger.ac.uk/cosmic). Префиксом идентификатора COSMIC должен быть "COSM" (например, COSM12979), как в стандартном профиле для генетики HL7 FHIR. COSMIC - это база данных для хранения и отображения информации о соматических мутациях и соответствующих деталей, содержащая информацию о раковых опухолях человека. COSMIC поддерживается Институтом сенгера от Независимого международного благотворительного фонда.
7.9 Классификация вариантов
7.9.1 Общая часть
В настоящее время используют два подхода для классификации генетических вариантов: один сфокусирован на патогенности, а другой - на клинической значимости возможной лекарственной терапии. Указанные два подхода имеют различные значения, поэтому лечебное учреждение может или сделать промежуточный выбор, какой из них будет использовать, или создать новую классификацию путем комбинирования двух имеющихся классификаций.
7.9.2 Классификация вариантов по патогенности
Настоящая классификация руководствуется рекомендациями ACMG в отношении стандартов по интерпретации и составлению отчетов по вариативности секвенирования: ревизия 2015 [17], система 5-категорийной классификации IARC [18] или рекомендаций College of American Pathologists [19], включающих патогенный, вероятно патогенный, неизвестного значения, вероятно доброкачественный, доброкачественный варианты.
Патогенный: классифицирован при заболевании более чем в трех несвязанных случаях с контрольными данными. Варианты локализованы в высококонсервативной области. Функциональные исследования или иные данные предполагают вредное воздействие на экспрессию генов.
Генетический вариант с доказательствами его патогенности: о вариантах сообщается в нескольких клинических наблюдениях с наличием или отсутствием контрольных данных. Функциональные исследования или иные данные (свидетельства) предполагают вредное воздействие на экспрессию генов.
Неизвестного значения: по данному варианту информация отсутствует или информация носит неполный и/или противоречивый характер.
Вероятно доброкачественный: о вариантах сообщается в малом количестве наблюдений, при небольшом количестве или при отсутствии контрольных данных. Вариант может быть неконсервативным и/или нести предсказательный (предрасполагающий) характер. Частота встречаемости выше ожидаемой в общей популяции, вне зависимости от распространенности или проявления заболевания. При этом варианты идентифицируются как функционально нормальные.
Доброкачественный: сообщается о высокой частоте в контрольных данных либо не связан с заболеванием. Вариант может быть неконсервативным и/или иметь предсказательный характер. При этом варианты идентифицируются как функционально нормальные.
7.9.3 Классификация вариантов по клинической значимости
В клинических условиях необходима альтернативная классификация для организации правильной терапии пациента [20]. В настоящем стандарте отредактирована следующая классификация: идентифицированный, вероятно идентифицированный, неопределенный и неидентифицированный.
Идентифицированный: выявляются клинически релевантные (реализуемые) варианты.
Вероятно идентифицированный: выявлены варианты с вероятной клинической значимостью.
Неопределенный: выявлены варианты с неопределенной клинической значимостью.
Неидентифицированный: не выявлено клинически значимых вариантов.
7.10 Рекомендуемая терапия
7.10.1 Общая часть
В отчет может быть внесена рекомендуемая терапия, например лекарственная терапия или клинические исследования для оказания помощи клиницистам.
7.10.2 Классификация вариантов по клинической значимости
Соответствующее лекарственное средство можно представить посредством идентификатора MPID (идентификатора лекарственного средства) или IMPID (экспериментального MPID) по ИСО 11615 IDMP (идентификация лекарственных средств). Классификационная система "The Anatomical Therapeutic Chemical (ATC)", поддерживаемая Центром ВОЗ по сотрудничеству в методологии статистических исследований (WHO Collaborating Centre for Drug Statistics Methodology), может быть использована в качестве альтернативы для представления классификации активных ингредиентов лекарственных средств. Система международных непатентованных названий (МНН) (International Nonproprietary Names, INN) улучшает идентификацию фармацевтических веществ или активных фармацевтических ингредиентов. Каждое МНН представляет собой уникальное название, признанное в глобальном масштабе и являющееся общественной собственностью. Если МНН существует, то МНН может быть альтернативно использовано как название лекарства.
Однако будет полезным в отдельных случаях, чтобы рекомендуемая цель терапии пациентов представлялась с использованием одинаковой схемы кодирования, уже применяемой в конкретной стране или имеющейся в электронном медицинском архиве, для того чтобы рекомендованная терапия и фактическая рецептура или курс химиотерапии семантически соответствовали в пределах регистрационной карты одного пациента, который, по всей вероятности, будет лечиться в местном, а не в глобальном масштабе. Таким образом, при наличии национального или частного стандарта его можно временно использовать. Примерами являются RxNorm, поддерживаемый Национальной медицинской библиотекой США (US. National Library of Medicine), или лекарственный стандарт KD (Korea Drug Code) Агентства "Korea Health Insurance Review and Assessment Service".
7.10.3 Информация по клиническим испытаниям
Клинические испытания определенного подбора ориентированных на варианты лекарств могут проводить для оказания помощи клиницистам. Информация по клиническим испытаниям должна быть представлена по базе данных ClinicalTrials.gov ID или номером по базе данных EudraCT. В качестве альтернативы можно использовать другой отечественный идентификатор регистратуры.
ClinicalTrials.gov представляет собой реестр поддерживаемых на федеральном или частном уровне клинических испытаний, проводимых в Соединенных Штатах Америки или в других странах. Если необходимо выявить варианты посредством соответствующих клинических испытаний, можно использовать ClinicalTrials.gov ID. Кроме того, может быть приведена информация по состоянию мишени, вмешательству, фазе и результатам исследований.
Пример - NCT00844506.
EudraCT (https://eudract.ema.europa.eu/) - это база данных по всем клиническим испытаниям, инициируемым в Европе. Номер EudraCT можно также использовать для представления соответствующих клинических испытаний.
7.10.4 Общепризнанные протоколы, относящиеся к варианту
Если найденные варианты классифицированы в качестве несущих практическую ценность для клинического применения, необходимо указать общепризнанные клинические протоколы.
Примеры - Протокол CPIC (Clinical Pharmacogenetics Implementation Consortium):
протокол NCCN (National Comprehensive Cancer Network) см. //www.nccn.org/professionals/physician_gls/f_guidelines.asp
7.10.5 Другие рекомендации
Могут быть включены другие рекомендации, например другие лабораторные испытания, включая другой заказ или параметры испытаний.
7.11 Дополнение
В случае дополнения его описание следует разместить в обязательном поле. Кроме того, можно включить причину создания дополнения.
8 Поля и их номенклатура дополнительных данных
8.1 Общая часть
Элементы данных для дополнительных полей в отчете о структурированном секвенировании, сфокусированные в основном на поддержке принятия клинического решения за счет добавления более подробной информации. Указанные поля могут также применяться для клинического испытания и прикладного исследования.
Таблица 3 - Элементы данных и их метаданные для заполнения дополнительных полей
Элементы данных | Метаданные (исходные) | ||
Анамнез | ICD | ||
Семейный анамнез/генеалогическая информация | HL7 v3 IG: семейный анамнез/генеалогическая интероперабельность | ||
Версия референсного генома | Genome Reference Consortium Human Genome release ID | ||
Информация по расовому типу генома | Текст | ||
Генетические | Символы и названия генов | HGNC | |
варианты | Информация о | Обозначение | HGVS |
вариативности | Эффекты вариативности | Текст | |
секвенирования | Идентификатор варианта секвенирования | Уникальный идентификатор базы данных | |
Версия HGVS | Номер версии HGVS | ||
Детальная | Дата клинического секвенирования | ИСО 8601 | |
информация | Индикаторы контроля качества | Числовой | |
по | Информация по | Глубина прочтения | Числовой |
секвенированию | восстановлению нуклеотидных | Референсная аллельная глубина | |
оснований | Альтернативная аллельная глубина | ||
Аллельная частота | |||
Генотип | |||
Информация по | Тип секвенатора | Текст | |
платформе секвенирования | Методы подготовки библиотеки | ||
Методы захвата мишени | |||
Тип прочтения | ENUM ("одностороннее", "парное") | ||
Длина прочтения | Текст | ||
Информация по платформе | Инструменты выравнивания | Текст | |
анализа | Инструменты вызова варианта | ||
Дополнительные инструменты | |||
Система координации хромосом | ENUM ("с 0 оснований", "с 1 основанием", "с 0 оснований, полуоткрытая") | ||
Аннотационные инструменты и базы данных | Текст | ||
Ссылки | Текст | ||
| |||
8.2 Анамнез
В отчет можно вносить анамнез обследуемого. При внесении в отчет анамнеза необходимо использовать терминологию соответствующего стандарта, например ICD для названий заболеваний или IDMP для лечебного средства.
8.3 Семейный анамнез/генеалогическая информация
Весь семейный анамнез или генеалогическая информация должны быть представлены посредством HL7 v3 Implementation Guide: Family History/Pedigree Interoperability, Release 1.
8.4 Версия референсного генома
Референсные последовательности являются исходным уровнем, с которого начинается отчет о варианте. При использовании различных референсных последовательностей вызовы вариантов тоже отличаются. Референсная последовательность должна быть представлена посредством Genome Reference Consortium Human Genome release ID or Locus Reference Genomic ID.
Референсные последовательности должны быть представлены посредством Genome Reference Consortium Human Genome release ID (//www.ncbi.nlm.nih.gov/projects/genome/assembly/grc/human/). При наличии исправления необходимо добавить номер исправления.
Пример - GRCh38 или GRCh37.c.13 (GRCh37 выпуск исправления 13).
LRG (Locus Reference Genomic) ID (//www.lrg-sequence.org/), поддерживаемый EBI, также можно использовать.
Пример - LRG_1.
8.5 Информация по расовому типу генома
После внесения в отчет информации по расовому типу генома необходимо указать набор референсных данных (ссылок) по данной информации, используя карты гаплотипов (//hapmap.ncbi.nlm.nih.gov/), 1000 геномов (//www.1000genomes.org/), комплект данных Exome Aggregation Consortium (ExAC) (//exac.broadinstitute.org/), базы данных по геномной агрегации (gnomAD) (//gnomad.broadinstitute.org/) или другой базы данных секвенирования, которая может выдать информацию о принадлежности к определенной расе.
8.6 Генетические варианты
Остающиеся генетические варианты, не занесенные в обязательную часть, могут быть перечислены в дополнительной части. Варианты необходимо вносить в отчет согласно 7.7. Однако в дополнительных полях следует указать версию HGVS.
Пример - HGVS Версия 15.11.
8.7 Детальная информация по секвенированию
Данные, автоматически создаваемые секвенатором, необходимо приводить в целях исследования. В настоящее время стандарта по этому вопросу не существует. Можно в качестве альтернативы использовать указание, применяемое в полях для общепринятой биоинформатики.
8.7.1 Дата клинического секвенирования
Дата клинического секвенирования представляет собой дату, на которую лаборатория-исполнитель составляет результаты секвенирования с использованием полученного образца. Как правило, лаборатория получает и обрабатывает достаточное количество образцов для работы секвенатора с целью оптимизации эффективности секвенатора. Кроме того, для выполнения процедуры секвенирования требуется не менее одного дня и более для обработки результатов. Дату оформляют по ИСО 8601 так же, как и остальные даты.
8.7.2 Показатели контроля качества
Можно указать соответствующие показатели контроля качества (QC). В отчет можно вносить общие показатели QC по биообразцу, показатели QC по всем вариантам или показатели QC по отдельно взятым вариантам на основании решения составителя отчета.
Пример - Выход секвенирования, число всех прочтений, длина (средняя) прочтения (bp), число прочтений, картированных к референсному геному (продуктивность картирования, %), N (основания не использовались для нуклеотидных оснований) основания (%), GC (%), Q20 (%), Q30 (%), точный охват (%)> 1x, точный охват (%)> 10x, точный охват (%)> 20x, точный охват (%)> 100x, средняя глубина и гомогенность.
Пример - GC=53,2%, AT=47,8%, Q30=94,4%, Q20=97,1%.
8.7.3 Информация по восстановлению нуклеотидных оснований
Информация по восстановлению нуклеотидных оснований создается при помощи программного обеспечения по восстановлению нуклеотидных оснований для идентификации относительно того, что нуклеотидную последовательность можно регистрировать. Выделяемые поля указаны ниже. Можно добавить дополнительные поля на основании решения составителя отчета.
8.7.3.1 Глубина прочтения
Среднее количество нуклеотидов, участвующих в части сборки, можно заносить в отчет как используемое в общепринятых областях биоинформатики.
Пример - 100x.
8.7.3.2 Референсная аллельная глубина
Аллельную глубину для референсной аллели можно заносить в отчет так же, как это принято при биоинформатической обработке.
Пример - 50x.
8.7.3.3 Альтернативная аллельная глубина
В отчет можно заносить глубину аллели для альтернативной аллели.
Пример - 50x.
8.7.3.4 Аллельная частота
В отчет можно заносить частоту альтернативной аллели в каждом локусе для каждого индивидуума.
Пример - 0,3, 30%.
8.7.3.5 Генотип
В отчет можно заносить пару вводимых аллелей в отдельном локусе.
Пример - AA, AC, AG, AT, CC, CG, CT, GG, GT, TT.
8.7.4 Информация по платформе секвенирования
Информацию по данным методикам секвенирования, включая платформу секвенирования, метод захвата и алгоритм выравнивания, необходимо изложить в текстовом формате.
8.7.4.1 Типы секвенаторов
Необходимо указать конкретный секвенатор, при помощи которого проводят секвенирование.
Пример - Illumina Hiseq 2500, Thermo Fisher Ion Torrent, Illumina MiSeq.
8.7.4.2 Методы подготовки библиотеки
Необходимо указать методы подготовки библиотеки секвенирования.
Пример - SureSelectXT.
8.7.4.3 Методы захвата мишени
Необходимо указать методы захвата экзома или целевой области.
Пример - Захват ампликона, пробы.
8.7.4.4 Тип прочтения
Необходимо указать тип прочтения секвенируемой области.
Пример - Одностороннее, парное.
8.7.4.5 Длина прочтения
Необходимо указать длину прочтения секвенируемой области.
Пример - 101 bp, 35-250 bp.
8.7.5 Информация по платформе анализа
Необходимо указать первичные, вторичные (при необходимости, третичные) источники анализа. Также необходимо упомянуть настройку для источников для подтверждения надежности вызова варианта.
Пример - GATK 3.5, CASAVA 1.7, Complete Genomics v2.2, Torrent Suite 5.0.2.
8.7.5.1 Инструменты выравнивания
Необходимо указать наименование инструмента выравнивания и его версии.
Пример - BWA-MEM 0.7.12.
8.7.5.2 Инструменты определения варианта последовательности
Необходимо указать инструмент определения варианта и его версию.
Пример - GATK 3.5, SAMTools 1.3.1.
8.7.5.3 Дополнительные инструменты
Пример - PICARD 1.9.3.
8.7.5.4 Система хромосомных координат
Позиция на хромосоме может начинаться с 0 или 1. Таким образом, система координации должна быть указана с началом в 0, с началом в 0 полуоткрытая или система координации с началом в 1.
8.7.5.5 Аннотационные инструменты и базы данных
Необходимо включить в отчет наименование аннотационных инструментов и источников баз данных, имеющихся в общедоступной форме или в частном владении.
Пример - ANNOVAR (2016Feb01), SnpEff, 4.3, Ensembl v74.
8.8 Ссылки
Вся информация должна быть изложена со ссылкой на соответствующие источники. Ссылки могут представлять собой опубликованные статьи или проверенную базу данных. Можно использовать унифицированный эталонный формат.
Приложение А
(справочное)
Примерная структура отчета о клиническом секвенировании
В настоящем приложении представлена композиция отчета о клиническом секвенировании с применением информативных значений.
Таблица А.1 - Резюмирующая часть
Поля | Значение | Представление | ||
Заказы клинического секвенирования | Код заказа по клиническому секвенированию | Код заказа | 14-RM-0000056 | 14-RM-0000056 |
Дата и время | Дата заказа | 2014-04-18 | 18 апреля 2014 | |
Дата отчета | 2014-04-25 | 25 апреля 2014 | ||
Информация об образце | 13-S-048435_A1 | 13-S-048435_A1 | ||
Информация об | Идентификаторы | 12345678 | 12345678 | |
обследуемом | ФИО | Gildong Hong | Gildong Hong | |
Данные о биоматериалах | Тип пробы | CEN | Некровеносная ткань | |
Генетические вариации | Символы и названия генов | HGNC:1097, BRAF | BRAF | |
Рекомендуемая терапия | Лекарственное средство | Vemurafenib, L01XE15 (стандарт АТС) | Vemurafenib | |
Общая интерпретация | Идентифицированный | Идентифицированный | ||
Примечание - Пример резюмирующей части.
| ||||
Таблица А.2 - Детальное содержание. Обязательные поля
Поля | Значение | Представление | ||
Заказ | Код заказа по | Код заказа | 14-RM-0000056 | 14-RM-0000056 |
клинического секвенирования | клиническому секвенированию | Информация о заказе секвенирования | OnkoPanel V2 (Целевой NGS для 505 генов, T/N пара) | OnkoPanel V2 (Целевой NGS для 505 генов, T/N пара) |
Дата и время | Дата заказа | 2014-04-14 | 14 апреля 2014 г. | |
Дата взятия образцов | 2014-04-17 | 17 апреля 2014 г. | ||
Дата получения | 2014-04-25 | 25 апреля 2014 г. | ||
Дата отчета | 2014-05-08 | 8 мая 2014 г. | ||
Информация об образце | 13-S-048435_A1 | 13-S-048435_A1 | ||
Информация | Идентификаторы | 12345678 | 12345678 | |
об обследуемом | ФИО | Gildong Hong | Gildong Hong | |
Дата рождения | 1947-04-29 | 29 апреля 1947 | ||
Пол | 1 | Мужской | ||
Национальность | 2040-04-01 | Кореец | ||
Данные об уполномоченном лице - заказчике клинического секвенирования | D060001 | Chulsoo Kim | ||
Лаборатория- | Базовая информация | АМС Genomic Pathology Lab | АМС Genomic Pathology Lab, 02-3010-8460 | |
Информация о составителе отчета | D110001 | Min Lee | ||
Информация о легально верифицированном лице по отчету о секвенировании | D030001 | Sejin Kim | ||
Сопутствующие заболевания и фенотипы | C34.90 | C34.90 Злокачественная опухоль неопределенной части неопреде- | ||
Данные о биоматериалах | Тип образца | CEN | Некровеносная ткань | |
Класс геномного источника в биоматериале | 2 | Соматический | ||
Состояния образца | Принят, процент опухолеобразования на тестируемой ткани, %: 90 | Принят, процент опухолеобразования на тестируемой ткани, %: 90 | ||
Примечание - Пример детализированной части (обязательные поля).
| ||||
Окончание таблицы А.2
Поля | Значение | Представление | ||
Генетические варианты | Символы и названия генов | HGNC:1097, BRAF | BRAF | |
Информация о вариативности секвенирования | Номенклатура HGVS | c.1799Т>A_p.V600E | c.1799Т>A_p.V600E | |
Эффекты вариативности | Замена (миссенс) | Замена (миссенс) | ||
Идентификатор варианта секвенирования | COSM476 | //cancer. | ||
Классификация | Патогенность | Патогенный | Уровень 1 | |
вариантов | Клиническая значимость | Замена (миссенс) | (патогенный, идентифициро- | |
Рекомендуемая терапия | Лекарственное средство | Vemurafenib, L01XE15 | Vemurafenib | |
Информация о клиническом испытании | NCT01791309 | Vemurafenib and Panitumumab Combination Therapy in Patients with BRAF V600E Mutated Metastatic Colourectal Cancer (https://clinicaltrials. | ||
Примечание - Пример детализированной части (обязательные поля).
| ||||
Таблица А.3 - Детальное содержание. Дополнительные поля
Поля | Значение | Представление | |
Анамнез | Не имеется | Не имеется | |
Семейный анамнез/генеалогическая информация | Не имеется | Не имеется | |
Версия референсного генома | GRCh37.p13 | GRCh37 выпуск исправления 13 (выпущено 28 июня 2013) | |
Информация по расовому типу генома | 1000 геномов | 1000 геномов | |
Генетические варианты | Символы и названия генов | HGNC:11998, ТР53 HGNC:17278, PNRC1 HGNC:30988, BNC2 | ТР53 |
Версия HGVS | Версия HGVS 15.11 | Версия HGVS 15.11 | |
Примечание - Пример дополнительной части (дополнительные поля).
| |||
Окончание таблицы А.3
Поля | Значение | Представление | ||
Детальная | Дата клинического секвенирования | 2014-05-04* | 4 мая 2017* г. | |
информация по секвенированию | Индикаторы контроля | Количество всех прочтений | 6950582 | 6,950,582 bp |
качества | Средний охват мишени | 121.2 | 121,2 X | |
% охваченных баз мишеней 100Х | 93.1 | 93,1% | ||
Информация по платформе | Тип секвенаторов | MiSeq | Illumina MiSeq | |
секвенирования | Методы подготовки библиотеки | SureSelectXT Reagent Kit, HSQ 96 | SureSelectXT Reagent Kit, HSQ 96 | |
Методы захвата мишени | SureSelect Biotinylated RNA library "Baits" (SureSelectXT Custom panel) | SureSelect Biotinylated RNA library "Baits" (SureSelectXT Custom panel) | ||
Тип прочтения | Парноконечное | Парноконечное | ||
Длина прочтения | 150 | 150 bp (75x2) | ||
Информация по платформе | Инструменты выравнивания | BWA | BWA-MEM 0.7.12 | |
анализа | Инструменты вызова варианта | SNV (Mutect2) | SNV (Mutect2) | |
Система координации хромосом | С одним основанием | С одним основанием | ||
Аннотационные инструменты и базы данных | VEP: предиктор геномных вариаций, версия 85 | VEP: предиктор геномных вариаций, версия 85 | ||
Референции | PMID: 12068308 PMID: 12198537 | Davies H, Bignell GR, Cox C, et al., Mutations of the BRAF gene in human cancer. Nature. 2002 Jun 27;417(6892): | ||
Примечание - Пример дополнительной части (дополнительные поля).
| ||||
________________
* Текст документа соответствует оригиналу. - .
Приложение В
(справочное)
Примерный формат отчета о клиническом секвенировании
Настоящее приложение демонстрирует пример отчета о клиническом секвенировании.
Аналитический отчет по мутациям
Клиническое секвенирование | Информация о пациенте | ||
Код заказа | 16-RM-00000001 | Идентификаторы | TBD160733 |
Наименование | Oncochaser | ФИО | Jungang Lee |
Дата заказа | 2016-10-18 | Дата рождения | 1970-12-11 |
Дата отчета | 2016-11-10 | Пол | Мужской |
Национальность | Кореец | ||
Диагноз | Рак легких | ||
Образец/информация о биоматериале | |||
Информация об образце | 13-S-048435_A1 | ||
Тип пробы | FFPE | ||
Генетические варианты | |
Символы и названия генов | HGNC: 1097, BRAF |
Классификация вариантов | |
Уровень 1 (патогенный, положительный) | |
Рекомендуемая терапия | |
Лекарственное средство | Vemurafenib |
Информация о клиническом испытании | NCT017991309 (подробную информацию по активным клиническим испытаниям см. на www.clinicaltrials.gov) |
Общая интерпретация | |
Положительный: обнаружена клинически действенная мутация | |
Детальная информация
Клиническое секвенирование | Информация о пациенте | ||
Код заказа | 16-RM-00000001 | Идентификаторы | TBD160733 |
Наименование | Oncochaser (Целевое NGS) | ФИО | Jungang Lee |
Дата заказа | 2016-10-18 | Дата рождения | 1970-12-11 |
Дата приема заказа | 2016-10-19 | Пол | Мужской |
Национальность | Кореец | ||
Дата отчета | 2016-11-10 | Заболевание и фенотип | Рак легких |
Образец/информация о биоматериале | |||
Информация об образце | 13-S-048435_A1 | ||
Тип пробы | FFPE | ||
Класс геномного источника | Соматический | ||
Характеристики образца | Приемлемый, процент опухолеобразования исследуемых тканей, %: 30 | ||
Терапевт, выдавший направление | |||
Медицинский центр ABC | Kim Chulsoo | ||
Лаборатория-исполнитель | |||
Лабораторная информация | TBI Clinical Genomic Lab | ||
Отчет подготовил | Heechul Kim | ||
Одобрил | Sejin Kim | ||
Генетические варианты | ||
Символы и названия генов | HGNC: 1097, BRAF | |
Информация о | Номенклатура HGVS | c.1799T>A_p.V600E |
вариативности секвенирования | Эффекты вариативности | Замена (Миссенс) |
ID варианта секвенирования | COSM476 | |
Классификация вариантов | ||
Уровень 1 (патогенный, положительный) | ||
Рекомендуемая терапия | ||
Лекарственное средство | Vemurafenib | |
Информация о клиническом испытании | NCT017991309 (подробную информацию по активным клиническим испытаниям см. на www.clinicaltrials.gov) | |
Семейный анамнез | Анамнез | Отсутствует |
Family history | Отсутствует | |
Версия референсного генома | GrCh37 | |
Информация по расовому типу генома | Отсутствует | |
Дополнительные генетические варианты | ||
Символы и названия генов | HGNC: 795, ATM | |
Информация о | Номенклатура HGVS (15.11) | c.-31+1129T>A |
вариативности | Эффекты вариативности | Интрон-вариант |
секвенирования | ID варианта секвенирования | Отсутствует |
Классификация вариантов | Уровень 4 (вероятно доброкачественный, отрицательный) | |
Рекомендуемая терапия | Лекарственное средство | Отсутствует |
Информация о клиническом испытании | Отсутствует | |
Общепринятые протоколы, относящиеся к варианту | Отсутствует | |
Другие рекомендации | Отсутствует | |
Символы и названия генов | HGNC: 11491, SYK | |
Информация о | Номенклатура HGVS (15.11) | c.154G>T_p.A52S |
вариативности | Эффекты вариативности | Замена (миссенс) |
секвенирования | ID варианта секвенирования | Отсутствует |
Классификация вариантов | Уровень 4 (вероятно доброкачественный, отрицательный) | |
Рекомендуемая терапия | Лекарственное средство | Отсутствует |
Информация о клиническом испытании | Отсутствует | |
Общепринятые протоколы, относящиеся к варианту | Отсутствует | |
Другие рекомендации | Отсутствует | |
Детальная информация по секвенированию | ||
Данные клинического секвенирования | 2016-10-25 | |
Показатели контроля | Количество всех прочтений | 12369192 |
качества | Средний охват мишени | 1839.36 Х |
% охваченных баз мишеней 30Х | 100% | |
Платформа | Тип секвенаторов | Hiseq2500 |
секвенирования | Методы подготовки библиотеки | SureSelectXT Reagent Kit |
Методы захвата мишени | SureSelect Biotinylated RNA library "Baits" (Custom panel) | |
Тип прочтения | Парные концы | |
Длина прочтения | 200 bp (100X2) | |
Информация по платформе анализа | Инструменты выравнивания | BWA-v0.7.12, PICARD-v1.92, GATK-v2.3-9 |
Инструменты вызова варианта | SNV, Indel (GATK-v2.3-9, Mutect-v1.1.4) | |
Дополнительные инструменты | Annotation | |
Система координации хромосом | 1-based | |
Аннотационные инструменты и базы данных | SnpEff v4.1g |
Библиография
[1] | International Cancer Genome Consortium, 2014 |
[2] | TARCZY-HORNOCH P., AMENDOLA L., ARONSON S.J., GARRAWAY L., GRAY S., GRUNDMEIER R.W. et al. A survey of informatics approaches to whole-exome and whole-genome clinical reporting in the electronic health record. Genet. Med. 2013, 15 (10), pp.824-832 |
[3] | ZERHOUNI E. The NIH Roadmap. Science. 2003, 302 (5642), pp.63-72 |
[4] | GOOD B.M., AINSCOUGH B.J., MCMICHAEL J.F., SU A.I., GRIFFITH O.L. Organizing knowledge to enable personalization of medicine in cancer. Genome Biol. 2014 Aug 27, 15 (8), p.438 |
[5] | ISO 25720:2009, Health informatics - Genomic Sequence Variation Markup Language (GSVML) [Информатика в здравоохранении. Язык разметки изменения геномной последовательности (GSVML)] |
[6] | HL7 Version 3 Domain Analysis Model: Clinical Sequencing, Release 1, 2014 //www.hl7.org/ |
[7] | FHIR DSTU2. 4.20.19 Standard Profile for Genetics, //www.hl7.org/FHIR/2015May/observation-genetics-cg-prf-1a.html |
[8] | SMART ON FHIR GENOMICS. //projects.iq.harvard.edu/smartgenomics |
[9] | ALTEROVITZ G., WARNER J., ZHANG P., CHEN Y., ULLMAN-CULLERE M., KREDA D. et al. SMART on FHIR Genomics: facilitating standardized clinico-genomic app. J. Am. Med. Inform. Assoc. 2015, 22 (6), pp.1173-1178 |
[10] | CDISC, Study Tabulation Model Implementation Guide: Pharmacogenomics/Genetics Version 1.0, 2015 |
[11] | REHM H.L., BALE S.J., BAYRAK-TOYDEMIR P., BERG J.S., BROWN K.K., DEIGNAN J.L. et al.; Working Group of the American College of Medical Genetics and Genomics Laboratory Quality Assurance Committee. ACMG clinical laboratory standards for next-generation sequencing. Genet. Med. 2013, 15 (9), pp.733-747 |
[12] |
|
[13] | SCHEUNER M.T., HILBORNE L., BROWN J., LUBIN I.M. members of the RAND Molecular Genetic Test Report Advisory Board. A report template for molecular genetic tests designed to improve communication between the clinician and laboratory. Genet. Test. Mol. Biomarkers. 2012, 16 (7), pp.761-769 |
[14] | VASSY J.L., MCLAUGHLIN H.M., MACRAE C.A., SEIDMAN C.E., LAUTENBACH D., KRIER J.B. et al. A one-page summary report of genome sequencing for the healthy adult. Public Health Genomics. 2015, 18 (2), pp.123-129 |
[15] | US FDA. Collection of Race and Ethnicity Data in Clinical Trials, //www.fda.gov/RegulatoryInformation/Guidances/ucm126340.htm#f14 |
[16] | LEHMANN S., GUADAGNI F., MOORE H., ASHTON G., BARNES M., BENSON E. et al.; International Society for Biological and Environmental Repositories (ISBER) Working Group on Biospecimen Science. Standard Preanalytical Coding for Biospecimens: Review and Implementation of the Sample PREanalytical Code (SPREC). Biopreserv. Biobank. 2012, 10 (4), pp.366-374 |
[17] | RICHARDS S., AZIZ N., BALE S., BICK D., DAS S., GASTIER-FOSTER J. et al.; ACMG Laboratory Quality Assurance Committee. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet. Med. 2015, 17 (5), pp.405-424 |
[18] | PLON S.E., ECCLES D.M., EASTON D., FOULKES W.D., GENUARDI M., GREENBLATT M.S. et al.; IARC Unclassified Genetic Variants Working Group. Sequence variant classification and reporting: recommendations for improving the interpretation of cancer susceptibility genetic test results. Hum. Mutat. 2008, 29 (11), pp.1282-1291 |
[19] | AZIZ N., ZHAO Q., BRY L., DRISCOLL D.K., FUNKE B., GIBSON J.S. et al. College of American Pathologists' laboratory standards for next-generation sequencing clinical tests. Arch. Pathol. Lab. Med. 2015, 139 (4), pp.481-493 |
[20] | FARWELL K.D., SHAHMIRZADI L., EL-KHECHEN D., POWIS Z., CHAO E.C., TIPPIN DAVIS B. et al. Enhanced utility of family-centered diagnostic exome sequencing with inheritance model-based analysis: results from 500 unselected families with undiagnosed genetic conditions. Genet. Med. 2015, 17 (7), pp.578-586 |
[21] | CAUDLE K.E., KLEIN T.E., HOFFMAN J.M., MULLER D.J., WHIRL-CARRILLO M., GONG L. et al. Incorporation of pharmacogenomics into routine clinical practice: the Clinical Pharmacogenetics Implementation Consortium (CPIC) guideline development process. Curr. Drug Metab. 2014, 15 (2), pp.209-217 |
[22] | ISO/TS 22220:2011, Health informatics - Identification of subjects of health care (Медицинская информатика. Идентификация субъектов медицинского обслуживания) |
[23] | ISO 8601, Data elements and interchange formats - Information interchange - Representation of dates and times (Элементы данных и форматы обмена. Обмен информацией. Представление дат и времени) |
[24] | ISO/TS 27527:2010, Health informatics - Provider identification (Информатика в здравоохранении. Идентификация провайдера) |
[25] | ISO 11615, Health informatics - Identification of medicinal products - Data elements and structures for the unique identification and exchange of regulated medicinal product information (Информатизация здоровья. Идентификация лекарственных средств. Элементы данных и структуры для уникальной идентификации и обмена информацией о регистрируемых лекарственных средствах) |
[26] | HL7 Version 2 Implementation Guide: Clinical Genomics; fully LOINC-Qualified Cytogenetics Model, Release 1 - US Realm |
[27] | HL7 CDA Implementation Guide for Genetic Testing Report (GTR) |
[28] | HL7 Fast Healthcare Interoperability Resource DSTU 2 Standard Profile for Genetics |
[29] | CDISC, Study Tabulation Model Implementation Guide: Pharmacogenomics/Genetics Version 1.0 |
УДК 004:61:006.354 | ОКС 35.240.80 |
Ключевые слова: здравоохранение, информатизация здоровья, метаданные, элементы данных, секвенирование | |
Электронный текст документа
и сверен по:
, 2019